HLA(human leukocyte antigen ,人类白细胞抗原)是人类的主要组织相容性复合体(MHC)的表达产物。通常也称为人类主要组织相容性复合物 (MHC),在染色体 6p21.3 上包含一个 4 Mb 的区域,其中包含 200 多个基因。
HLA根据不同的基因位点分为三大类型:I型,II型和III型。
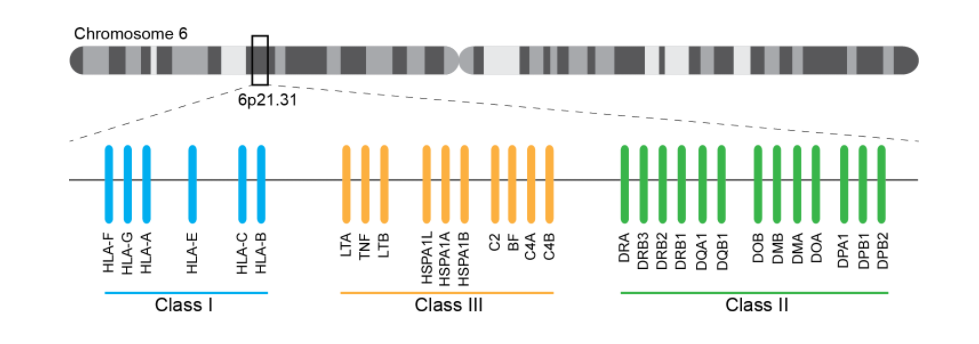
I 类分子存在于大多数细胞类型中,并存在从癌细胞、被感染细胞(病毒)的细胞内环境中衍生出来的内源性多肽。I 类分子中的结合肽呈递给 CD8+T 细胞,CD8+T 细胞会杀死受感染/受损的细胞。II 类分子主要存在于抗原呈递细胞上,并将抗原呈递给 CD4+ T 辅助细胞。III 类包含细胞因子基因和补体系统成分,它们在免疫反应中也发挥重要作用。
HLA-I型基因,包括了HLA-A,HLA-B,HLA-C等经典的抗原基因,还有一些假基因;HLA-II 型基因,其编码产物都是双链蛋白质,包括DR,DP,DQ等基因,HLA-III型基因,包含了C2,C4等补体基因,还包括其他一些基因。
HLA 复合体是整个人类基因组中多态性最强的区域,最新的统计信息如下:

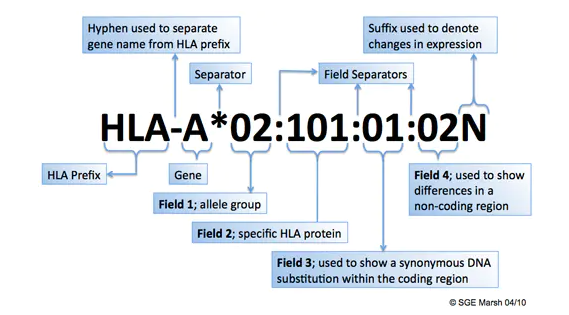
HLA命名方式:
有众多基于全外显子组测序(WES)或全基因组测序(WGS)的HLA分型算法及工具,其中Optitype(基于整数线性规划的HLA基因分型算法)是运用的相对比较多的一款对HLA-I类分型工具。
1) Optitype 环境部署
- Python 2.7(Python3.6也可以,推荐用conda部署)
- RazerS 3.4
- SAMtools 1.2
- HDF5 1.8.15
- CPLEX 12.5 其他一些线性规划解释器 (GLPK, CBC, …推荐用linux安装,windows会缺少这一部分软件库比较麻烦)
python需要的依赖模块:
- NumPy 1.9.3
- Pyomo 4.2
- PyTables 3.2.2
- Pandas 0.16.2
- Pysam 0.8.3
- Matplotlib 1.4.3
- Future 0.15.2
2)安装
目前有两种方式来安装 Optitype :
1、基于docker
如果有docker,那就比较简单了,直接:docker pull fred2/optitype
然后执行:docker run -v /path/to/data/folder:/data/ -t fred2/optitype -i input1 [input2] (-r|-d) -o /data/
2)从源码直接安装,推荐用docker搭建环境
conda create -n optitypeconda install -n optitype -c bioconda optitype
如果安装python3.6的建议先用conda创建python环境,然后安装依赖
3)测试
修改config.ini.example文件里面的参数,重命名为config.ini
先使用razers3对测序数据进行mapping(由于 razers3 是将所有的数据读取到内存,需要注意)
razers3 -i 95 -m 1 -dr 0 -o fished_1.bam /path/to/OptiType/data/hla_reference_dna.fasta sample_1.fastq然后用samtools处理bam
samtools bam2fq fished_1.bam > sample_1_fished.fastq注意:双端测序数据两个fq都需要分别执行上面操作
用OptiType进行分型:
DNA数据:
python OptiTypePipeline.py -i ./test/exome/NA11995_SRR766010_1_fished.fastq ./test/exome/NA11995_SRR766010_2_fished.fastq --dna -v -o ./test/exome/RNA数据:
python OptiTypePipeline.py -i ./test/rna/CRC_81_N_1_fished.fastq ./test/rna/CRC_81_N_2_fished.fastq --rna -v -o ./test/rna/注意HLA的数据库可以在:ftp://ftp.ebi.ac.uk/pub/databases/ipd/imgt/hla/下载更新
关于HLA I类基因的分析软件 OptiType 就介绍到这了。
Q&A
如果运行遇见“ImportError HDFStore requires PyTables No module named tables”之类的错误建议查看相应的python环境,可以尝试如下解决方案:
pip install --upgrade tables
参考资料:
1.https://github.com/FRED-2/OptiType
2.Szolek, A, Schubert, B, Mohr, C, Sturm, M, Feldhahn, M, and Kohlbacher, O (2014). OptiType: precision HLA typing from next-generation sequencing data Bioinformatics, 30(23):3310-6.
3.https://stackoverflow.com/questions/25980018/importerror-hdfstore-requires-pytables-no-module-named-tables
4.https://sciscogenetics.com/pages/technology.html
 浙公网安备 33010802011761号
浙公网安备 33010802011761号