我们常用的免疫浸润分析工具主要有CIBERSORT和xcell, CIBERSORT 目前引用比较多;两者的思路是完全不同的。 CIBERSORT 是通过去卷积方法来分析细胞组成成分,而 xcell 是通过整合了基因富集分析的优势(基于ssGSEA),可以评估多大64种细胞类型,涉及多个适应性和先天免疫细胞、造血祖细胞、上皮细胞和细胞外基质细胞,本文简单介绍xCell工具的使用。
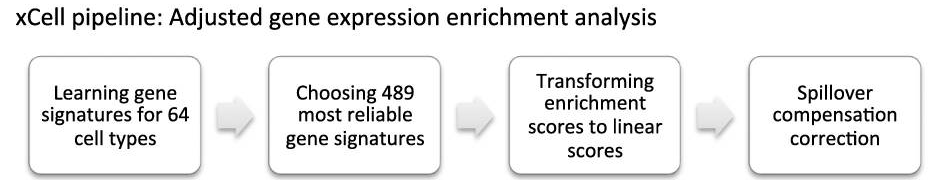
xCell首先利用用机器学习算法提取了64种免疫细胞和基质细胞的signature,然后对待检测bulkRNA数据先用ssGSEA算法计算样本在每个细胞类型signature的富集分数,然后将样本中各种细胞类型的富集分数转换为细胞类型分数,最后将相关的细胞类型分数进行校正,该方法适用于基因表达谱和传统RNA-seq数据,如果是单细胞数据推荐该团队的另一个工具https://github.com/dviraran/SingleR 。
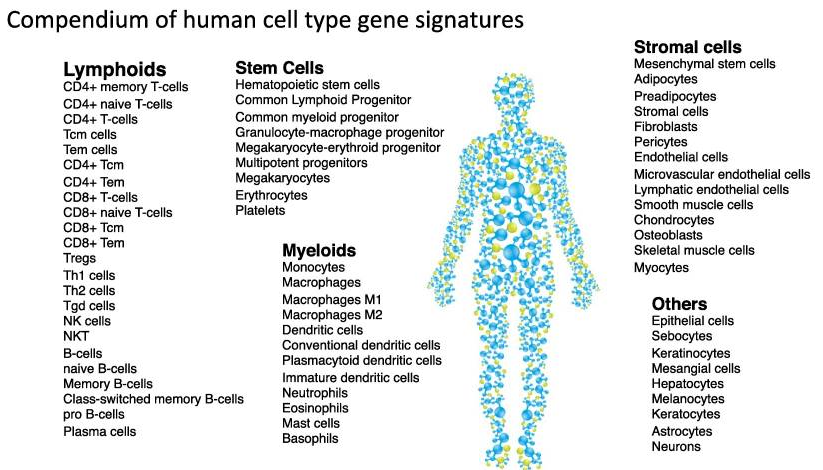
xCell有网页版和R包两种形式使用:
1)网页版
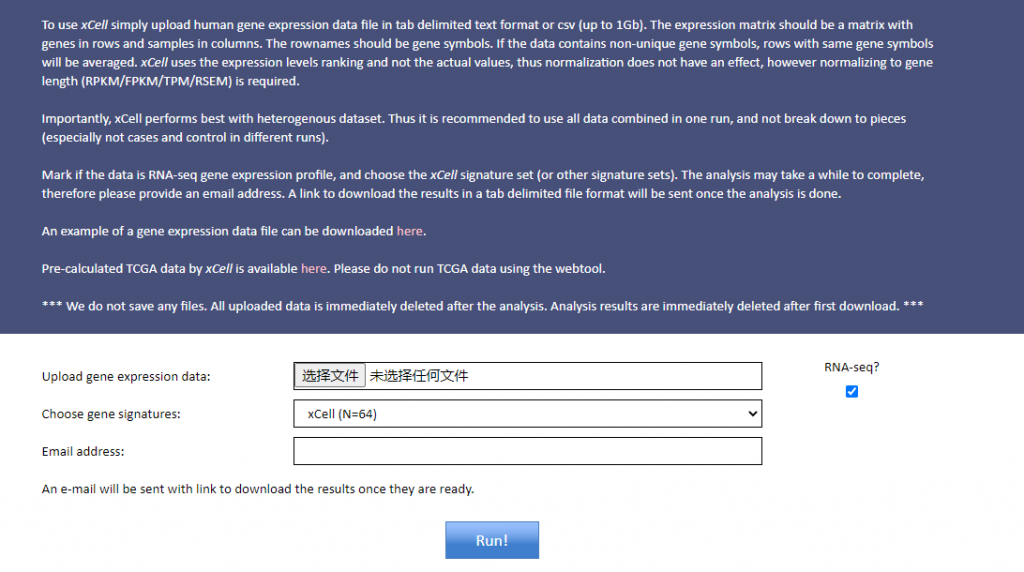
只需要上传基因表达数据,填写好自己的邮箱地址,结果生成后会直接发送到邮箱,非常方便没有复杂的操作。
2)R 包版本
R包:https://github.com/dviraran/xCell
安装:
devtools::install_github('dviraran/xCell')使用方法:
library(xCell) exprMatrix = read.table(expr_file,header=TRUE,row.names=1, as.is=TRUE) xCellAnalysis(exprMatrix)
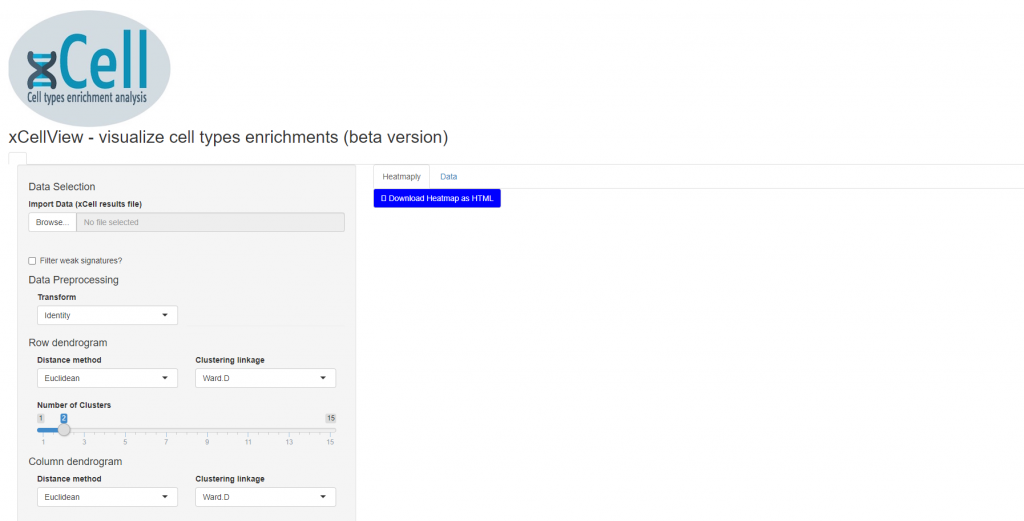
同时作者还提供了一个数据可视化工具:https://comphealth.ucsf.edu/app/xcellview/
参考资料:
1.https://xcell.ucsf.edu/
2.Aran D, Hu Z, Butte AJ. xCell: digitally portraying the tissue cellular heterogeneity landscape. Genome Biol. 2017;18(1):220. Published 2017 Nov 15. doi:10.1186/s13059-017-1349-1

 浙公网安备 33010802011761号
浙公网安备 33010802011761号