网络图绘制方法有很多,biocGraph也是基于igraph构建的一个更加易用的网络图R包,可以直接用各种统计图表来代替各个网络节点,呈现更多的信息给读者,也为生物学的可视化展示的复杂性带来便捷。
关于igraph的介绍,本文不再赘述,可以参考《利用igraph绘制网络图》学习。
下面将简单介绍biocGraph的用法和示例:
# 安装
if (!requireNamespace("BiocManager", quietly = TRUE))
install.packages("BiocManager")
BiocManager::install("biocGraph")一个示例:
library(biocGraph)
# 设置随机数,便于重复
set.seed(123)
# 定义网络,可以参考igraph
V <- letters[1:10]
M <- 1:4
g1 <- randomGraph(V, M, 0.2)
counts = matrix(rexp(numNodes(g1) * 4), ncol = 4)
g1layout <- agopen(g1, name = "foo")
# 定义点的呈现方式,此处采用官方文档介绍的饼图展示
makeNodeDrawFunction <- function(x) {
force(x)
function(node, ur, attrs, radConv) {
nc <- getNodeCenter(node)
# 饼图属性设置
pieGlyph(
x,
xpos = getX(nc),
ypos = getY(nc),
labels = NA,
radius = getNodeRW(node),
col = rainbow(4)
)
# 饼图文本内容
text(
getX(nc),
getY(nc),
paste(signif(sum(x), 2)),
cex = 0.5,
col = "white",
font = 1
)
}
}
# 绘制网络图
drawFuns <- apply(counts, 1, makeNodeDrawFunction)
plot(g1layout, drawNode = drawFuns, main = "Example Pie Chart Plot")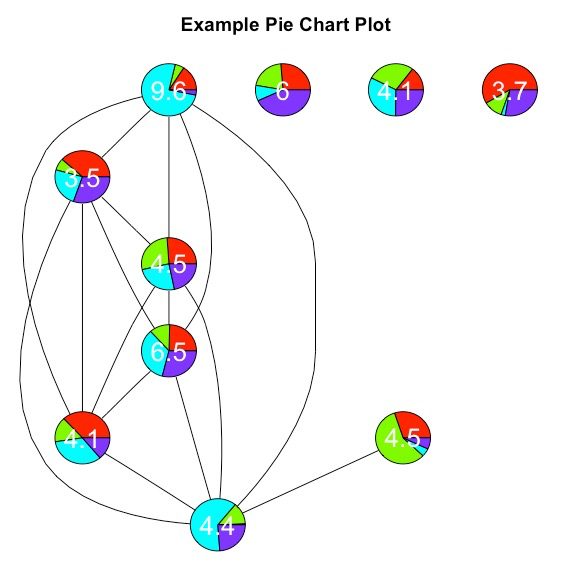
参考资料:
1.http://bioconductor.org/packages/release/bioc/html/biocGraph.html
 浙公网安备 33010802011761号
浙公网安备 33010802011761号