Biopython(http://www.biopython.org)目的是为使用和研究生物信息学的研究者提供了一个工具集,包括了一些模块、脚本等内容。目前,Biopython致力于通过创造可重复利用的模块及类,使得Python在生物信息学中的研究、应用变得更加简单、容易。
Biopython的特点包括解析各种生信文件文件(如:BLAST, Clustalw, FASTA, Genbank…),访问一些在线的数据库(NCBI,Expasy…),和一些程序的接口(Clustalw, DSSP,MSMS…)等等,同时也衍生出如BioPerl,BioRuby,BioJava扩展。
| 入门 | 帮助文档 | 贡献 |
|---|---|---|
| 下载 Biopython | 教程 (PDF) | 目前正在进行 |
| README文档 | wiki文档 | 通过Github |
| Cookbook (示例) | 通过Google | |
| Q&A文档 | 提交bugs |
功能介绍:
- 将生物信息学文件解析为Python可利用的数据结构,包括对以下格式的支持:
- Blast的输出结果
- Clustalw
- FASTA
- GenBank
- PubMed 和 Medline格式
- ExPASy文件
- SCOP,包括 ‘dom’ 和 ‘lin’ 文件
- UniGene
- SwissProt
- 被支持的文件可以通过记录来重复或者通过字典界面来索引
- 和常见的生物信息学在线数据库交互:
- NCBI – Blast,Entrez 和 PubMed
- ExPASy – Swiss-Prot 和 Prosite ,包括Prosite搜索
- 常见生物信息学软件的接口:
- 单机版Blast
- Clustalw
- EMBOSS
- 可以处理序列、ID和序列特征的标准序列类
- 对序列实现常规操作的工具,如序列翻译,转录和权重计算等
- 利用k最近邻接、Bayes或SVM对数据进行分类
- 通过创建和处理替换矩阵的方法来进行序列比对
- 分发并行任务
- 其GUI的程序可以进行基本的序列操作
- 详细的文档
- 与BioSQL集成,同样也被BioPerl和BioJava项目支持
案例:
1)翻译CDS条目为FASTA文件
from Bio.SeqRecord import SeqRecord
from Bio import SeqIO
def make_protein_record(nuc_record):
"""Returns a new SeqRecord with the translated sequence (default table)."""
return SeqRecord(seq = nuc_record.seq.translate(cds=True), \
id = "trans_" + nuc_record.id, \
description = "translation of CDS, using default table")
proteins = (make_protein_record(nuc_rec) for nuc_rec in \
SeqIO.parse("coding_sequences.fasta", "fasta"))
SeqIO.write(proteins, "translations.fasta", "fasta")2)序列GC%含量作图
from Bio.SeqUtils import GC
import pylab
gc_values = sorted(GC(rec.seq) for rec in SeqIO.parse("ls_orchid.fasta", "fasta"))
pylab.plot(gc_values)
pylab.title("%i orchid sequences\nGC%% %0.1f to %0.1f" \
% (len(gc_values),min(gc_values),max(gc_values)))
pylab.xlabel("Genes")
pylab.ylabel("GC%")
pylab.show()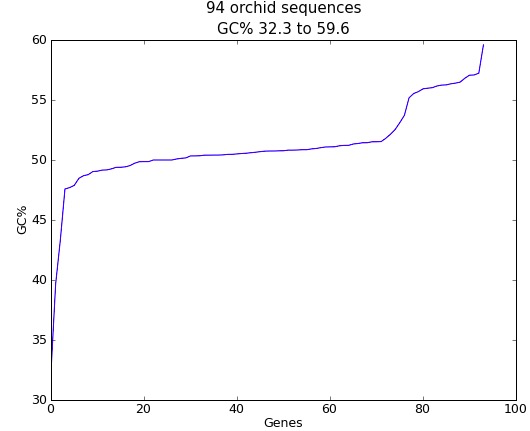
还有很多示例,此处就不再介绍了,更多内容可以参考下面资料。
参考资料:
1.http://biopython.org/DIST/docs/tutorial/Tutorial.html
2.https://biopython-cn.readthedocs.io/zh_CN/latest/index.html

 浙公网安备 33010802011761号
浙公网安备 33010802011761号