K邻近算法最初由 Cover和Hart于1968年提出,是一个理论上比较成熟的方法,也是目前比较简单和成熟的算法,在生命科学领域中应用相对广泛。其主要思想是:如果一个样本在特征空间中的K个最相邻的样本中大多数都属于某一个类,则该样本也属于这一类,并具有该类别上样本的特性。K邻近具体实现主要是通过两两样本之间的距离,如果两者距离较其他样本之间更近,那么则判断为同一类,一般选取欧式距离或曼哈顿距离来计算其远近。

R中有许多包都可以实现knn算法,如:class、DMwR、kknn包等都可以进行快速的KNN算法实现。
下面我们通过 kknn 做简单演示说明:
library(kknn)
library(caret)
set.seed(123456)
# 标准化
iris[, 1:4] <-
as.data.frame(lapply(iris[, 1:4], function(x) {
(x - min(x)) / (max(x) - min(x))
}))
# 随机选择70%数据作为训练集
train <-
sample(1:nrow(iris), size = nrow(iris) * 0.7, replace = FALSE)
# 训练集
train_data <- iris[train,]
# 测试集
test_data <- iris[-train,]
# 调用knn
# 可选的核函数:"rectangular"、"triangular"、"epanechnikov" 、 "biweight" 、
# "triweight"、 "cos"、 "inv"、 "gaussian"、 "rank"、"optimal"
# k默认等于7
pre <-
kknn(Species ~ .,
train_data,
test_data,
distance = 1,
kernel = "triangular")
fit <- fitted(pre)
# 计算混淆矩阵
confusionMatrix(table(fit, test_data$Species))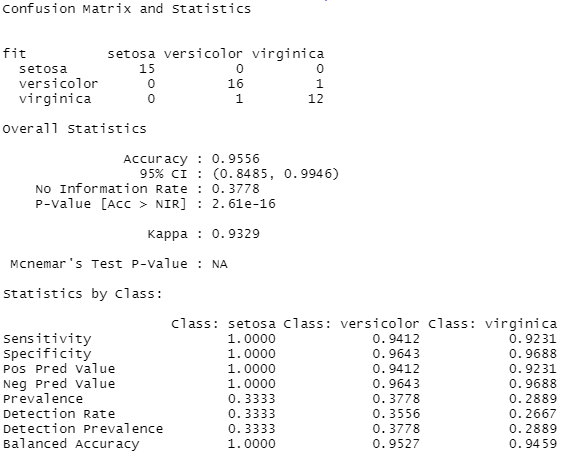
对于knn算法来说,k值确定是比较困难的。一般我们根据准确率来简单判定最佳k值,我们尝试所有可能K值并计算准确率:
# 选取最佳K值
iris_acc <- numeric()
for (i in 1:100) {
predict <- kknn(
Species ~ .,
train_data,
test_data,
distance = 1,
k = i,
kernel = "triangular"
)
fit <- fitted(predict)
iris_acc <- c(iris_acc, confusionMatrix(table(fit, test_data$Species))$overall['Accuracy'])
}
# 绘图
plot(
1 - iris_acc,
type = "l",
ylab = "Error Rate",
xlab = "K",
main = "Error Rate for Iris With Varying K"
)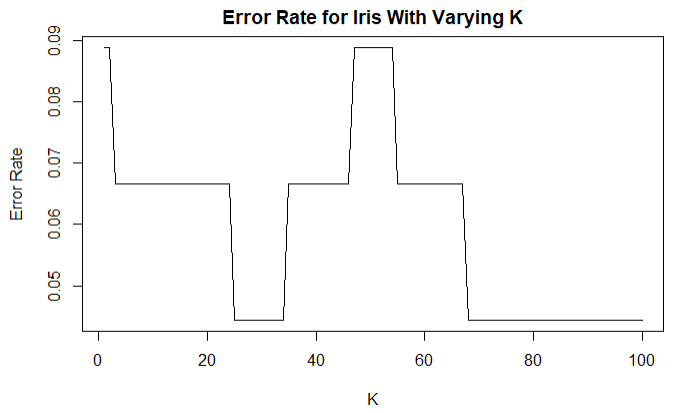
K值选择是没有统一的定论, 在实际应用中,K值一般取一个比较小的数值,同时也可以像采用交叉验证法来选择最优的K值。
总体来说KNN算法是相对比较简单易用的方法,值得ML初学者学习。
参考资料:
1.https://kevinzakka.github.io/2016/07/13/k-nearest-neighbor/
2.http://www.learnbymarketing.com/tutorials/k-nearest-neighbors-in-r-example/
3.https://www.edureka.co/blog/knn-algorithm-in-r/

 浙公网安备 33010802011761号
浙公网安备 33010802011761号