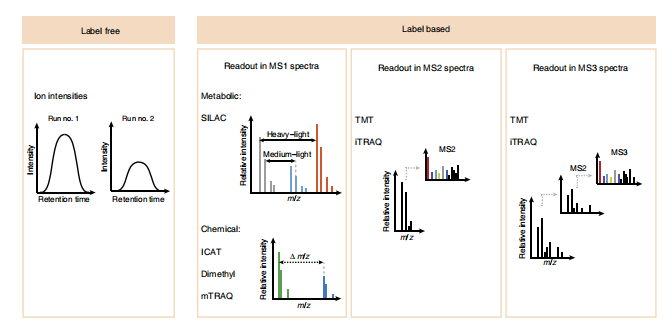
MaxQuant是基于质谱(Ms)的蛋白质组学DDA数据采集模式下的数据定量分析金标准,现在广泛应用于DDA数据分析,支持LFQ和Label的数据分析。
MaxQuant 支持Windows和Linux(Linux版基于mono)
下载地址:访问
基本工作原理:
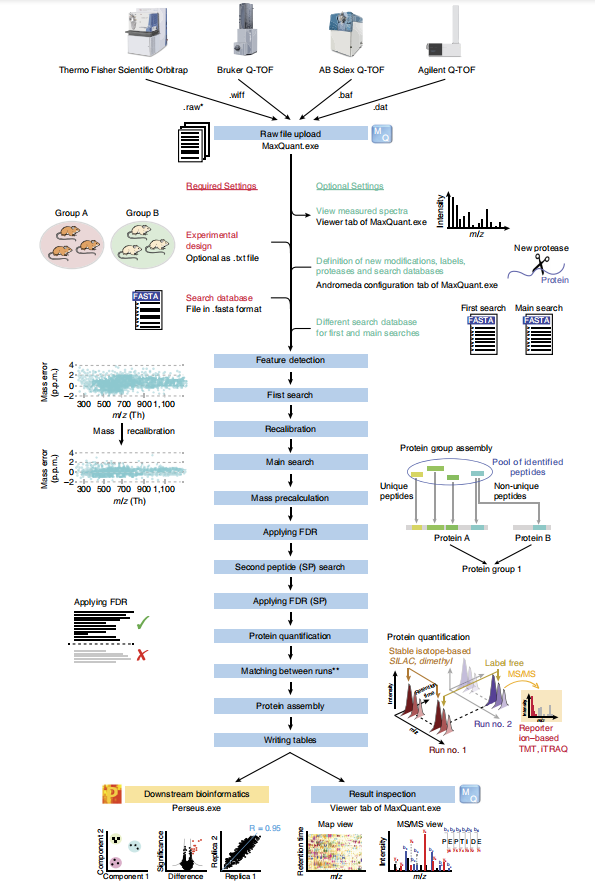
支持市面上几乎所有仪器设备的原始Raw文件,其搜库引擎采用了Andromeda,针对不同的方法采用自定义评分方案,对每种特定的碎片化技术的多肽鉴定进行优化,最后利用target-decoy搜索策略估计FDR和控制肽段鉴定种的假阳性。
下面简单介绍常规的DDA中Label-free的分析步骤:
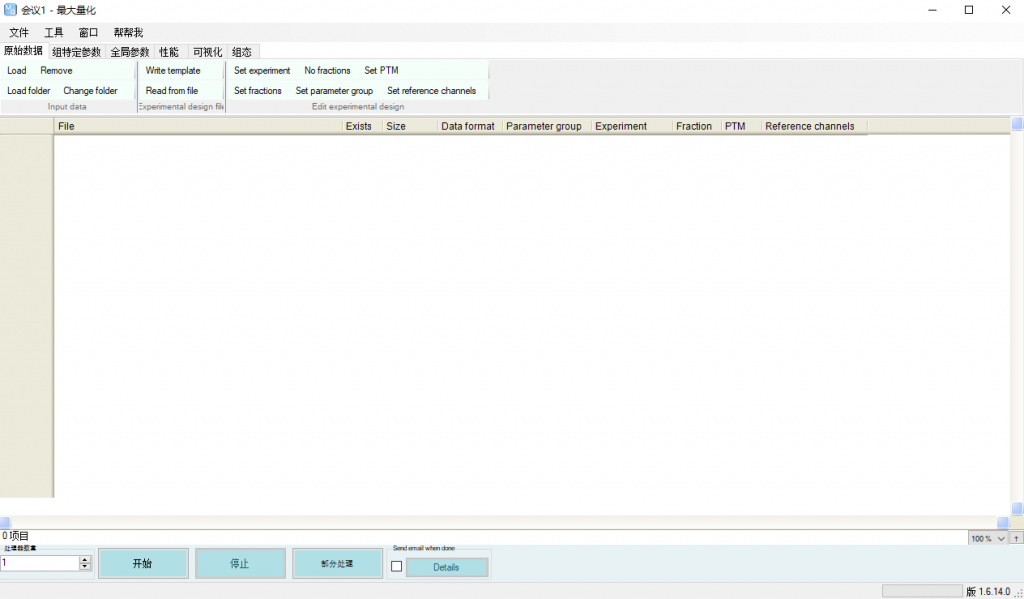
1)打开 MaxQuant 选择导入数据(可以选择导入文件或者文件夹)
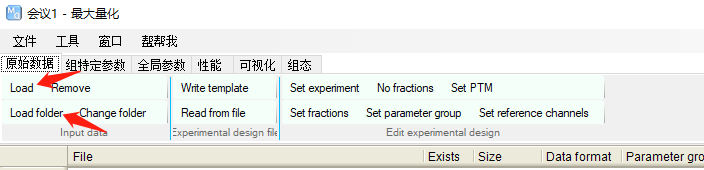
2)设置分组等信息
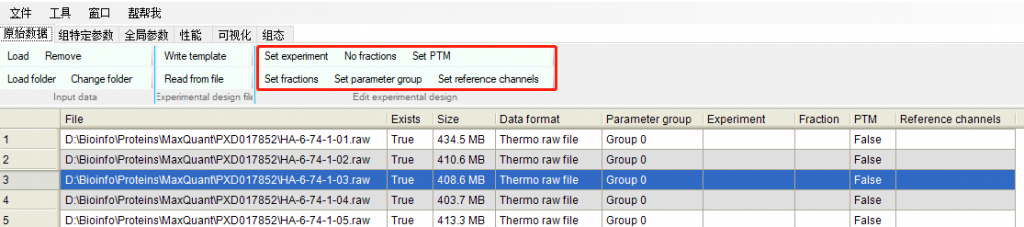
3)特定配置参数

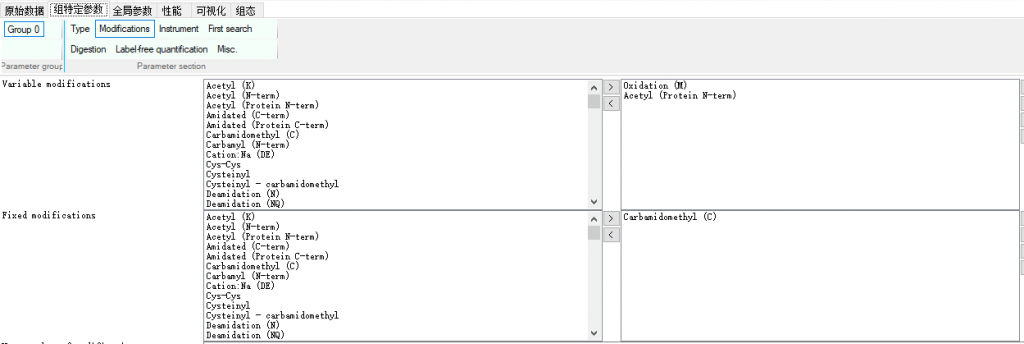
根据你的实验修饰参数, 消化酶切(一般我们用Trypsin/P)等参数设置
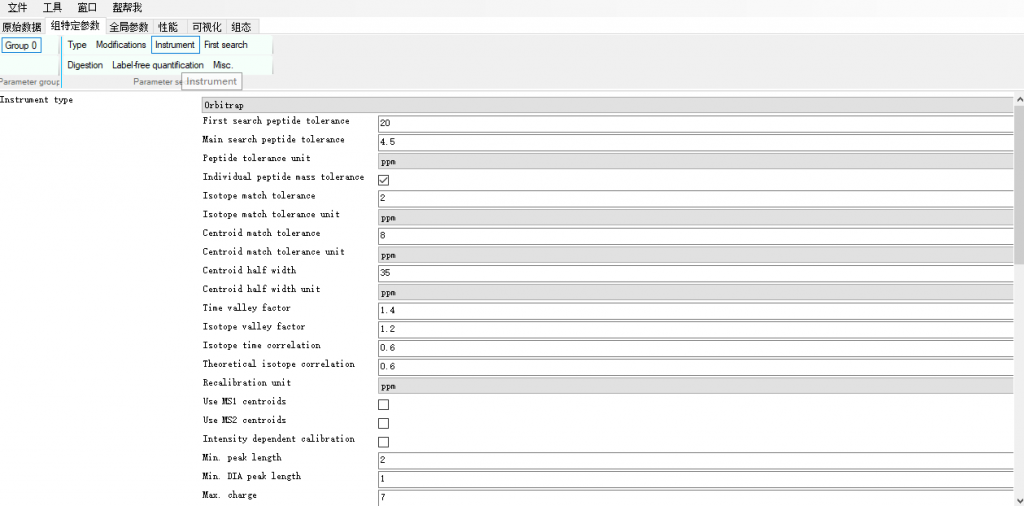
根据你使用的仪器配置Instrument参数
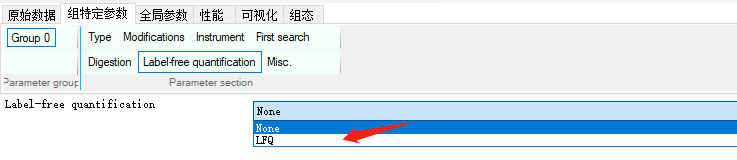
如果是无标定量,在 Label-free quantification选择LFQ
3)设置全局参数
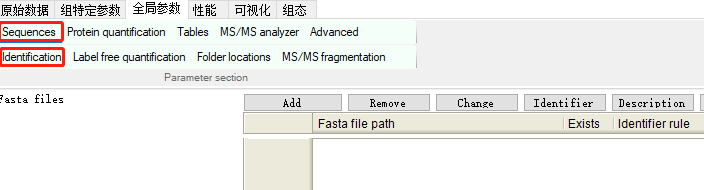
此处主要介绍Sequences和identification参数的配置,第一个主要是配置fasta文件,第二个是需要配置FDR等修改阈值(默认是0.01)
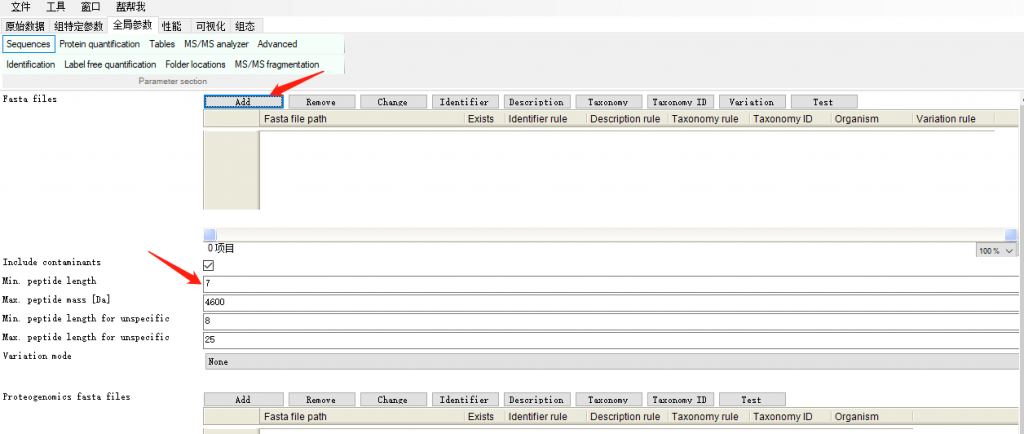
一般我们是从uniport上下载蛋白的fasta序列,点击Add即可添加,后面的参数像肽段最小长度等一般来说默认就可以了,也可以设置的更加宽松一点,这个根据你的需要来修改。
剩下主要是鉴定打分了,此处默认是0.01,相对比较保守的一个阈值,有人也设置到0.05,提高一些低信号肽的鉴定,但这样也会影响增加假阳性的可能,当然这之间的取舍还是根据你的实验目的来。大部分后期还会进行验证,所以这块的把控还是按照实验设计进行,0.01和0.05目前都是被认可的阈值。
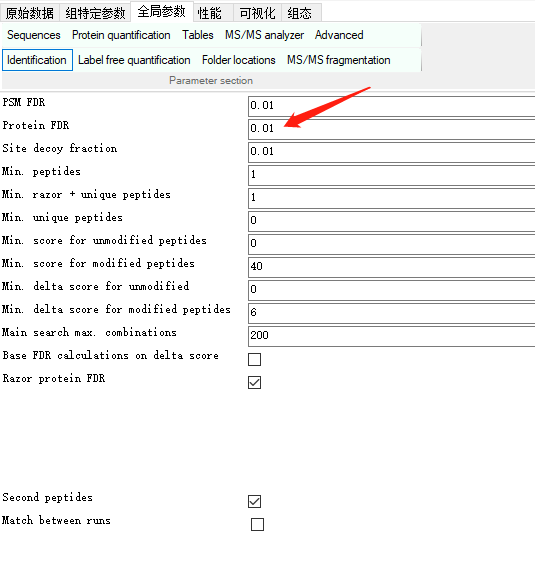
其他的高级参数像 target-decoy生成方式等,大家可以自行探索。
4)其他设置
剩下的三个页面和分析基本上没什么大的关系了。
性能主要是执行后的一个日志展示;
可视化主要是对Raw文件数据的展示:

组态主要是软件参数,像修饰等的修改添加等:
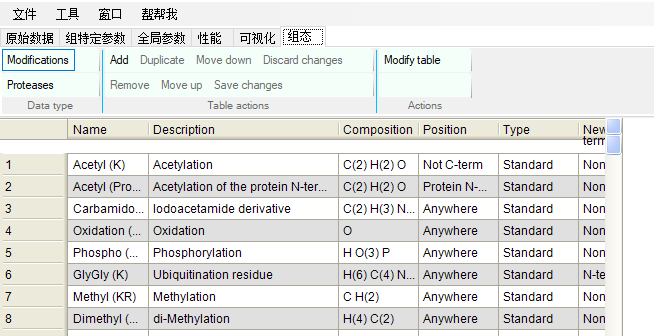
5)运行分析
配置完成后,我们根据自己的电脑配置设置处理器数量,如电脑8核设置8即可(个人建议,如果电脑除了跑 MaxQuant 还有其他任务,建议设置数量是你的电脑核数减1):

整个分析过程介绍结束了,大家也可以根据 Nat Protoc文章详细了解所有的参数信息。
注意:安装时提示缺失.NET core x.x 组件可以前往https://dotnet.microsoft.com/zh-cn/download/dotnet 下载。
参考资料:
1.http://coxdocs.org/doku.php?id=maxquant:start
2.Tyanova, S., Temu, T. & Cox, J. The MaxQuant computational platform for mass spectrometry-based shotgun proteomics. Nat Protoc11, 2301–2319 (2016).
3.Cox, J. et al. Andromeda: a peptide search engine integrated into the
MaxQuant environment. J. Proteome Res. 10, 1794–1805 (2011).
 浙公网安备 33010802011761号
浙公网安备 33010802011761号