Kraken2是微生物测序数据分析中较为常用的软件,与其他同类的软件相比,不仅速度快准确性也很高。作者在2014年发布了kraken,但是kraken对内存要求很大程度的限制了部分用户使用,kraken2优化了这一问题而且对下游分析上面也更加方便。
下图是kraken和kraken2的官方的比较图:
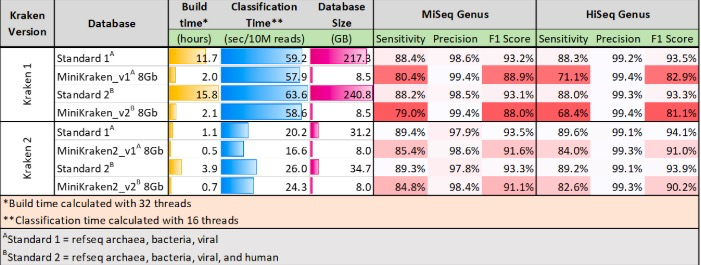
尤其在内存占用方面已经大大的降低了,下面简单介绍其安装和使用方法。
1)安装部署:
下载源代码:https://github.com/DerrickWood/kraken2
# 从github下载
git clone https://github.com/DerrickWood/kraken2.git
# 或wget下载并解压
wget https://github.com/DerrickWood/kraken2/archive/master.zip
unzip master.zip
# 选择安装的位置,开始安装,最后出现“Kraken 2 installation complete.”说明安装成功
KRAKEN2_DIR=~/kraken2
./install_kraken2.sh $KRAKEN2_DIR
# 添加到环境变量
cp $KRAKEN2_DIR/kraken2{,-build,-inspect} $HOME/bin或者可以通过conda安装,https://anaconda.org/bioconda/kraken2
conda install -c bioconda kraken22)配置数据库
kraken2可以自己构建数据库也可以下载构建好的数据库。
下载官方构建好的数据库(ftp://ftp.ccb.jhu.edu/pub/data/kraken2_dbs/),或者第三方构建好的数据库( https://github.com/rrwick/Metagenomics-Index-Correction)
- MiniKraken2_v1_8GB: (5.5GB) 8GB Kraken 2 Database built from the refseq bacteria, archaea, and viral libraries.
- MiniKraken2_v2_8GB: (5.5GB) 8GB Kraken 2 Database built from the Refseq bacteria, archaea, and viral libraries and the GRCh38 human genome
- Kraken 2 16S Greengenes 13_5 DB (72.1 MB)
- Kraken 2 16S RDP 11.5 DB (168 MB)
- Kraken 2 16S Silva 132 DB (117 MB)
- Kraken 2 16S Silva 138 DB (112 MB)
- GTDB_r89_54k linkA collection of database files for use with Centrifuge, Kraken 1, or Kraken 2 that can be used to classify metagenomes using the GTDB_389_54k index. More information and details at: https://github.com/rrwick/Metagenomics-Index-Correction
- Maxikraken2 and Kraken2-microbial databases. These databases are maintained by LomanLab. More information at the link provided.
标准数据库构建方法:
kraken2-build --standard --threads 24 --db $DBNAME其中DBNAME是指定数据库存储的位置,此步下载数据超过50GB的数据,数据库在创建期间将使用超过100 GB的磁盘空间。默认下载5种数据库:古菌archaea、细菌bacteria、人类human、载体UniVec_Core、病毒viral。
kraken2可以下载如下:”archaea”, “bacteria”, “plasmid”, “viral”, “human”, “fungi”, “plant”, “protozoa”, “nr”, “nt”, “env_nr”, “env_nt”, “UniVec”, “UniVec_Core”数据库,也可以指定下载数据库:
kraken2-build --download-library nr --threads 24 --db $DBNAME
# 也可以添加单个序列到数据库,如下格式:
# >sequence16|kraken:taxid|32630 Adapter sequence
# CAAGCAGAAGACGGCATACGAGATCTTCGAGTGACTGGAGTTCCTTGGCACCCGAGAATTCCA
kraken2-build --add-to-library chr1.fa --db $DBNAME
# 添加完成后需要构建数据库,建立索引
raken2-build --build --db $DBNAME3)序列分类
# 利用上一步建好的数据库进行分类
kraken2 --db $DBNAME seqs.fa4)环境变量设置
KRAKEN2_NUM_THREADS:设置默认线程数
KRAKEN2_DB_PATH:设置默认数据库位置如:export KRAKEN2_DB_PATH="/home/user/my_kraken2_dbs:/data/kraken2_dbs:"就可以直接调用相应路径下的数据库,kraken2 --db mainDB sequences.fa
参考资料:
1.https://github.com/DerrickWood/kraken2/wiki/Manual
2.https://genomebiology.biomedcentral.com/articles/10.1186/s13059-019-1891-0
3.https://ccb.jhu.edu/software/kraken2/index.shtml?t=downloads
 浙公网安备 33010802011761号
浙公网安备 33010802011761号