通常单一的组学不能提供足够的信息来深入了解生物系统,但是我们可以通过组合多个组学数据分析来获得对系统更加全面的了解。
mixOmics 软件包是在许多生物学研究中开发和验证的一整套多变量方法,来帮助我们更深入地进行多组学的研究。mixOmics提供了广泛的多元方法来探索和整合生物学数据集,尤其着重于变量选择。多变量方法非常适合大型的组学数据集,其中变量(例如基因,蛋白质,代谢产物)的数量远大于样本(病人,细胞,小鼠)的数量。它们通过使用工具变量(“组件”)减小数据维度的吸引人的特性,这些变量定义为所有变量的组合。然后使用这些组件来可视化输出,可以让我们更好地了解不同数据集之间的关系和相关结构。
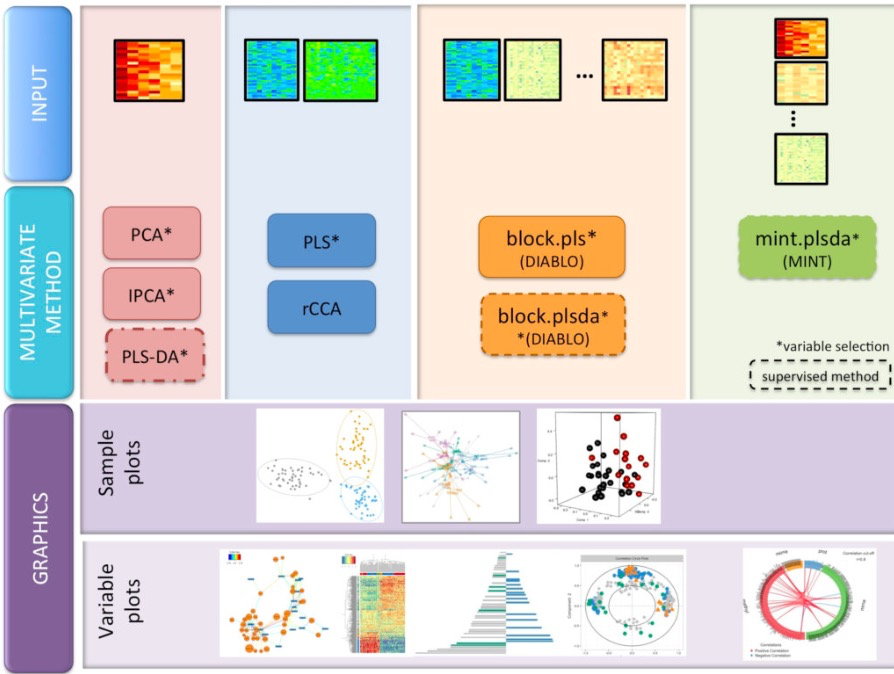
包含
- 统计方法:PCA/IPCA/CCA/PLS/PLS-DA/MixMC/MINT/DIABLO
- 可视化:散点图/相关性网络图/聚类图/相关性圈图/AUC等
安装:
if (!requireNamespace("BiocManager", quietly = TRUE))
install.packages("BiocManager")
BiocManager::install("mixOmics")教程:
- Principal Component Analysis (PCA)
- PLS – Discriminant Analysis (PLS-DA)
- Projection to Latent Structure (PLS)
- Multi-block Discriminant Analysis with DIABLO
总体来说为多组学数据研究提供了便利。
参考资料:
1.http://mixomics.org/
2.http://bioconductor.org/packages/release/bioc/vignettes/mixOmics/inst/doc/vignette.html
3.https://mixomicsteam.github.io/Bookdown/

 浙公网安备 33010802011761号
浙公网安备 33010802011761号