Granatum是基于shiny的一个单细胞RNA-seq(scRNA-seq)可视化在线分析工具。该工具从原始数据矩阵开始对scRNA-seq数据进行分析处理,包含了全面的分析模块:质量控制/标准化,聚类,差异基因表达/富集分析,蛋白质网络相互作用可视化和细胞伪时间构建等分析内容。
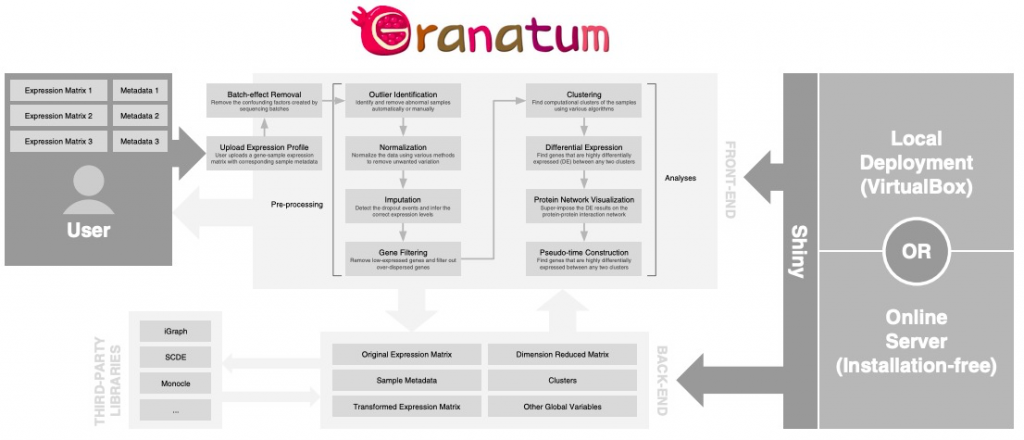
Granatum相比其他的单细胞分析流程来说有很多优势,附上官方比较信息:

在线地址:http://granatum.dcmb.med.umich.edu:8102/
也可以通过github上的源代码构建本地Granatum。
下面从官方的网站一步步简单介绍其使用方法:
1)上传数据

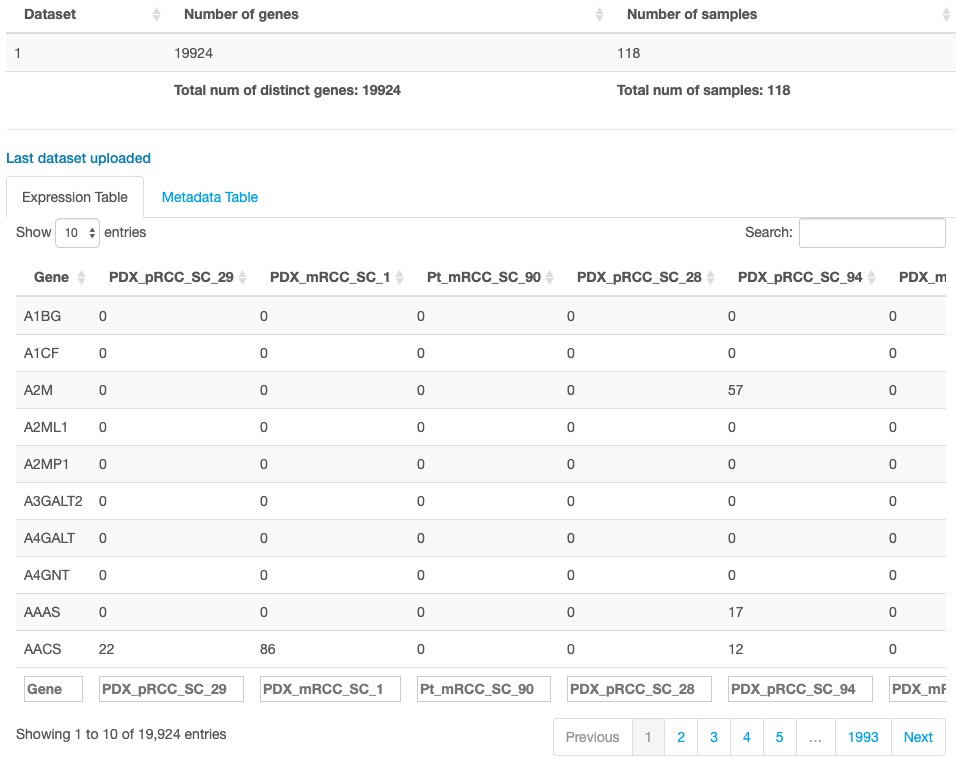
数据格式如下,此处我们下载官方演示数据进行上传测试:

上传完成后,会显示上传数据信息,如果确认数据没有问题点击“submit”提交即可:
2)去除批次效应,默认是用combat来去除
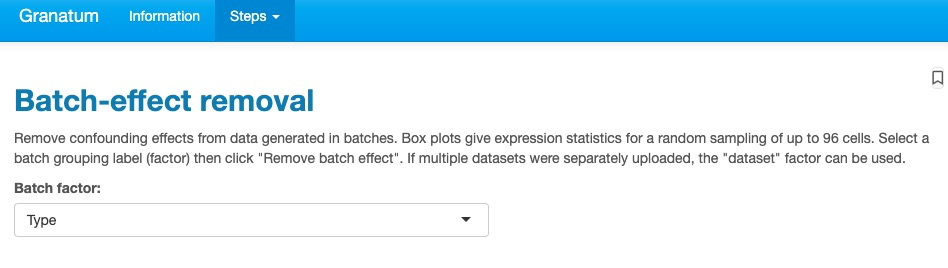
选中产生批次效应的来源,一般是不同的仪器、不同的时间、不同人做的实验等等,然后点击
完成之后点击“submit”进行下一步。
3)删除离群值/异常值
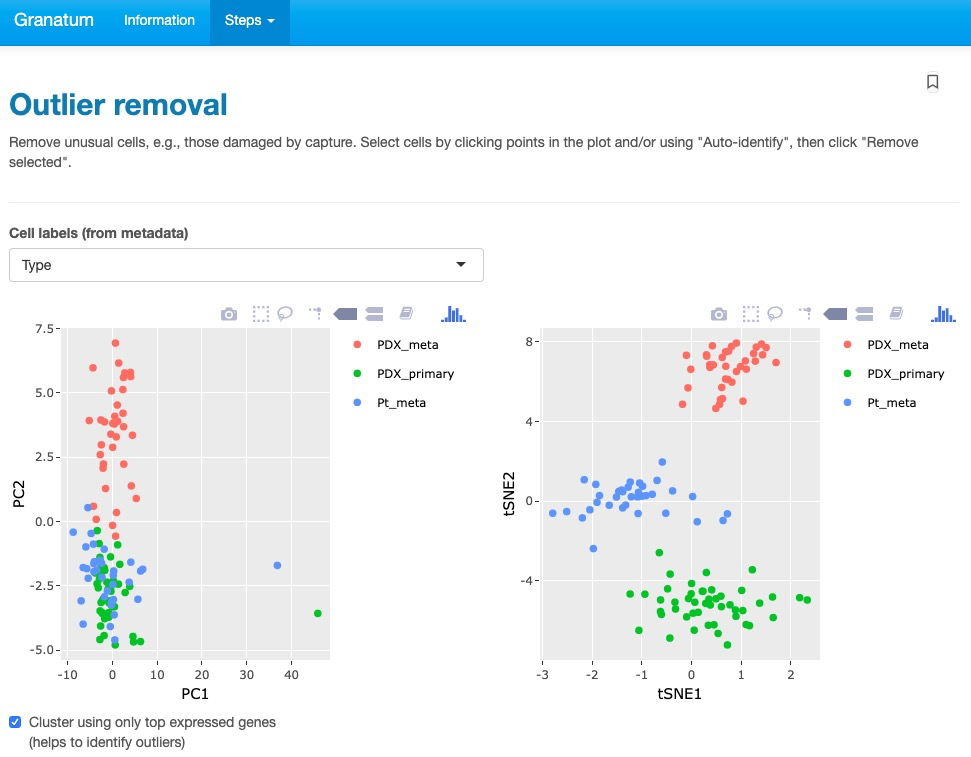
有一下三种方法供选择:
4)数据标准化

提供了如下几种方法来对数据进行标准化:

一般我们使用Quantile方法进行标准化。
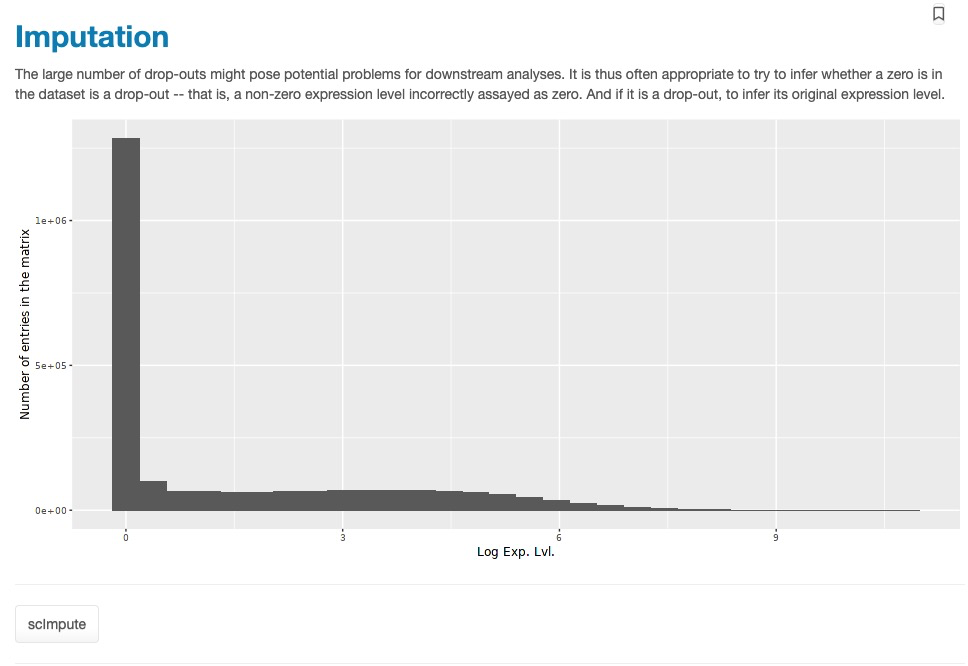
5)数据填充,一般是对缺失值或者0值需要进行填充或者过滤
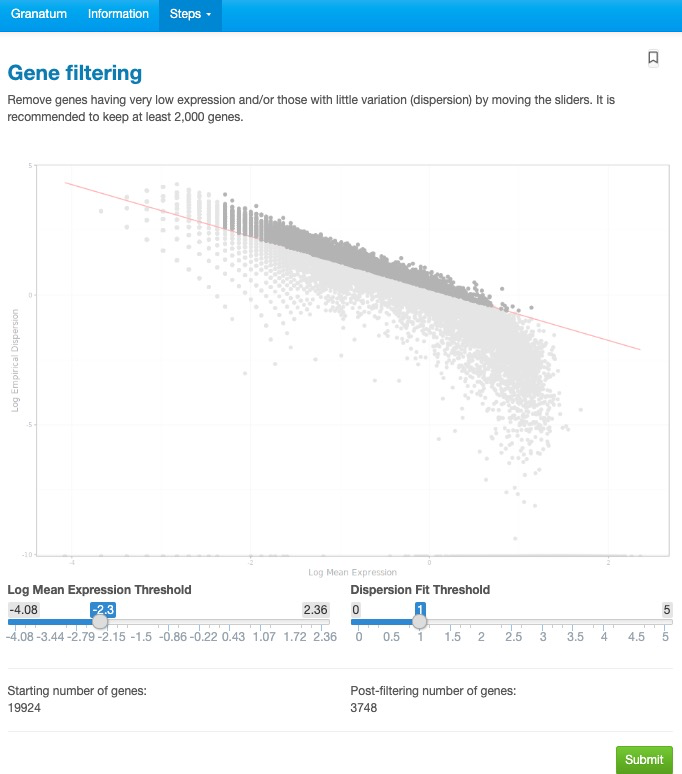
6)过滤低表达的基因
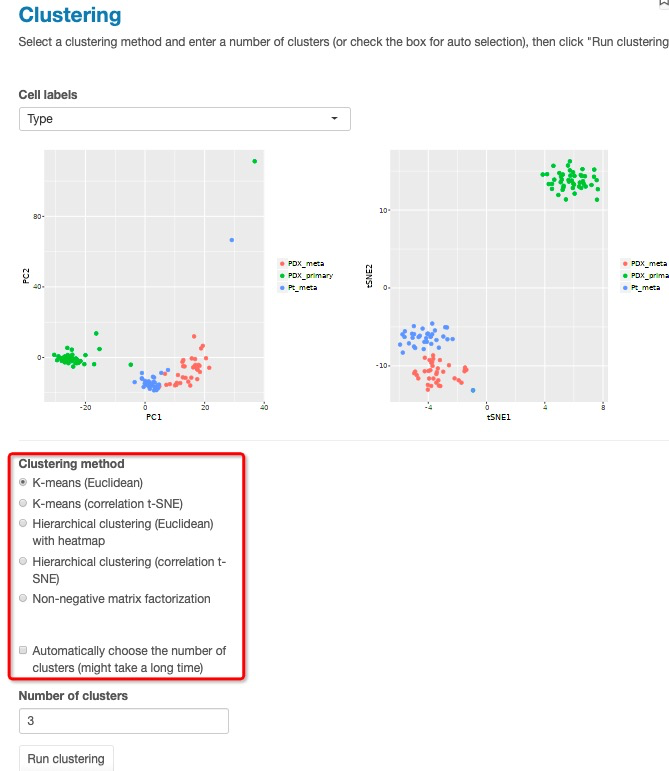
7)聚类分析
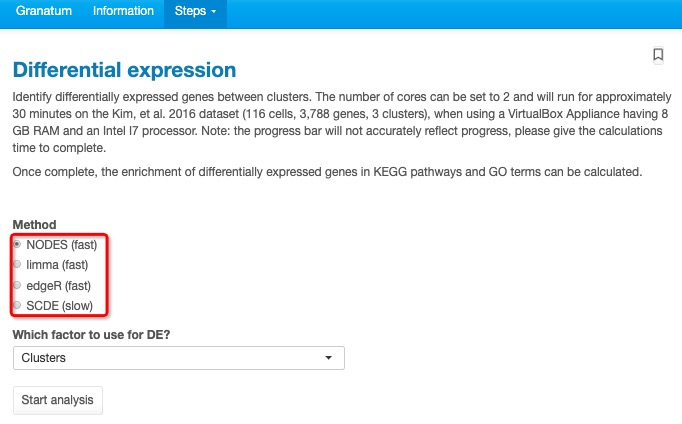
8)差异基因分析
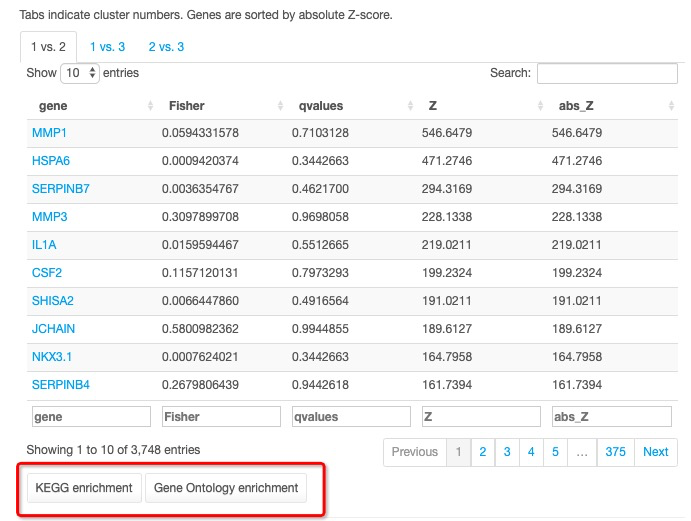
差异分析完成后可以进行下游的GO/KEGG分析等
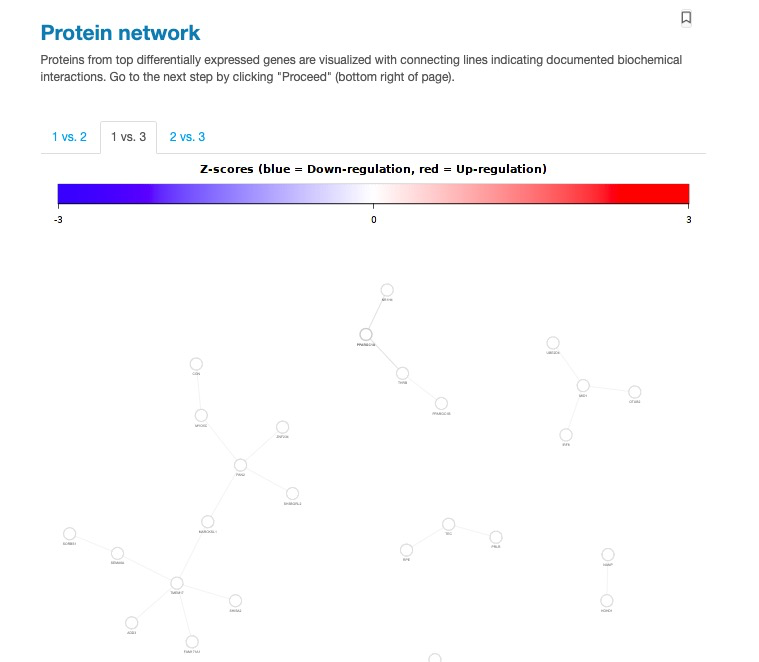
9)蛋白互作分析
10)伪时间序列分析

Granatum基本上包含了单细胞分析的整个流程,整体操作下来也是很便捷的,对用户操作也很友好。
参考资料:
1.http://granatum.dcmb.med.umich.edu:8102/
2.https://github.com/lanagarmire/Granatum
3. Zhu, Xun et al. “Granatum: A Graphical Single-Cell RNA-Seq Analysis Pipeline for Genomics Scientists.” Genome Medicine 9.1 (2017)

 浙公网安备 33010802011761号
浙公网安备 33010802011761号
陈浩
✅云上构建指引:
1.创建一台Ubuntu 18 VM主机
2.SSH登陆VM主机
3.运行machine_setup.sh ( https://github.com/lanagarmire/granatum ,或者可以单独运行这些命令)
4.运行:Rscript installDependencies.r
5.验证所有R环境是否安装正确:Rscript load_dependencies.r
6.下载源代码git clone https://github.com/lanagarmire/granatum
7.cd granatum1/server
npm install .
node app.js
8.确保云Firewall 和 Ubuntu Firewall的Granatum相应端口已经被放行
建议:
Ubuntu: sudo apt-get install libcap2-bin; sudo setcap cap_net_bind_service=+ep /usr/local/bin/node
Fedora: sudo yum install libcap-devel; sudo setcap cap_net_bind_service=+ep /usr/local/bin/node
R的默认R_MAX_NUM_DLLS调整:echo “R_MAX_NUM_DLLS=500” > ~/.Renviron