eggNOG数据库(http://eggnog5.embl.de/#/app/home)是NCBI的COG数据库(Clusters of Orthologous Groups of proteins,可以对预测蛋白进行功能注释、归类以及蛋白进化分析)的扩展,它收集了了5090个生物(真核生物、代表性细菌和古菌)和2502个病毒的全基因组蛋白序列(其中原核生物的一般称为COG数据库;真核生物的一般称为KOG数据库)。
eggNOG不仅提供了本地化软件,也提供了网页工具进行注释。
1)网页版
网址:http://eggnog-mapper.embl.de/
网页端操作简单,按照提示操作提交任务,任务完成后会通过邮件通知用户结果,eggNOG整个工作流程如下:
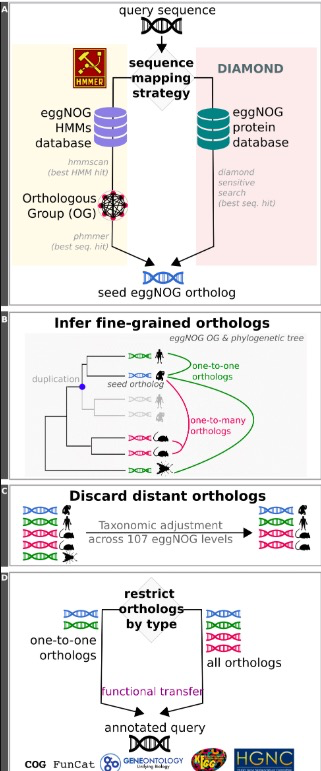
2)本地安装
需要安装python2.7和DIAMOND软件(建议用conda安装)
需要硬盘空间大小:
- eggNOG注释数据库:~40GB
- eggNOG序列fasta文件:~10GB
软件下载:
- eggnog:https://github.com/eggnogdb/eggnog-mapper
- eggnog数据库:http://eggnogdb.embl.de/download/
- diamond:https://github.com/bbuchfink/diamond/
- HMMER3:http://www.hmmer.org/download.html
- Anaconda3:https://repo.anaconda.com/archive/
软件安装完成后,需要加入环境变量。
下载数据库,用download_eggnog_data.py即可,脚本会自动将注释数据下载到data/目录下面。
部署完成后可以用以下命令测试:
# 测试命令
python emapper.py -i test/p53.fa --output p53_maNOG -m diamond
# 查看帮助信息
python emapper.py --help输出注释文件包含下面列信息:
1. query_name
2. seed eggNOG ortholog
3. seed ortholog evalue
4. seed ortholog score
5. Predicted taxonomic group
6. Predicted protein name
7. Gene Ontology terms
8. EC number
9. KEGG_ko
10. KEGG_Pathway
11. KEGG_Module
12. KEGG_Reaction
13. KEGG_rclass
14. BRITE
15. KEGG_TC
16. CAZy
17. BiGG Reaction
18. tax_scope: eggNOG taxonomic level used for annotation
19. eggNOG OGs
20. bestOG (deprecated, use smallest from eggnog OGs)
21. COG Functional Category
22. eggNOG free text description参考资料:
1.http://eggnog-mapper.embl.de/
2.https://github.com/eggnogdb/eggnog-mapper/wiki/eggNOG-mapper-v2#Installation

 浙公网安备 33010802011761号
浙公网安备 33010802011761号