提到曼哈顿图,做基因组的小伙伴肯定不陌生了,其通常用于全基因组关联研究(GWAS)以显示重要的SNP。目前R生态圈中绘制曼哈顿图的R包也有很多,如:qqman、ggman、CMplot、manhplot、ggplot2等。
目前用的比较多的像CMplot和ggplot2使用频率相对比较高,主要原因可能是其定制化程度比较大。除了qqman其他几个包都是基于ggplot2构建。
曼哈顿图本质上是一个散点图或者抖动图,X 轴为染色体编号,Y轴为该位点相关的统计显著性 Pvalue 值或者 FDR 值等
本文以基本的qqman介绍曼哈顿图的绘制:
library(qqman)
library(RColorBrewer)
# 第一列为SNP的名字,
# 第二列CHR为所在染色体(注意:染色体数据类型是整型,如果非数字类型可以修改成数字,后续可以修改坐标轴替换),
# 第三列BP为染色体上所在位置
# 第四列是对应的P值,你也可以用其他有意义的值
data("gwasResults")
# qqman使用的是plot函数绘制,理论上所有支持plot的参数都适用
manhattan(
gwasResults,
main = "Manhattan Plot",
chr = "CHR",
bp = "BP",
snp = "SNP",
p = "P",
# 点的大小,作者函数里面和添加SNP注释有冲突,可以选择性展示
# cex = 0.5,
# 坐标轴文字大小
cex.axis = 0.7,
# 自定义颜色,如果数据有20组,定义5组颜色,颜色会重复4次
col = brewer.pal(5, "Set3"),
# 例如此处我重新定义染色体标号
chrlabs = c(1:20, "X", "Y"),
# 高亮显示
highlight = c("rs3056", "rs3054"),
# 添加参考线
suggestiveline = -log10(0.01),
genomewideline = -log10(0.05),
# 根据 p-value 阈值对SNP进行注释
annotatePval = 0.01,
# 选择前5注释
annotateTop = 5
)
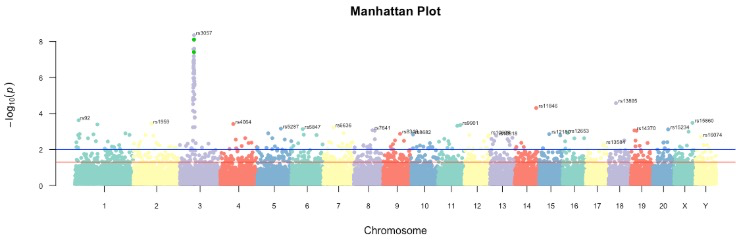
本文仅做一个抛砖引玉,相对而言,用ggplot2系列更方便定制。
参考资料:
1.https://github.com/YinLiLin/CMplot
2.https://github.com/stephenturner/qqman
3.https://github.com/cgrace1978/manhplot
4.https://github.com/drveera/ggman
 浙公网安备 33010802011761号
浙公网安备 33010802011761号