EBImage包括常用的图像处理和分析功能,像图像预处理,图像拼接等,本文以给细胞分裂染色图片添加边界为例,简单演示其用法:
# 安装S
BiocManager::install("EBImage")1)载入示例
# 载入包
library(EBImage)
# 加载示例,细胞分裂的一个案例
nuc = readImage(system.file('images', 'nuclei.tif', package='EBImage'))
cel = readImage(system.file('images', 'cells.tif', package='EBImage'))
# 读取图片
cells = rgbImage(green=1.5*cel, blue=nuc)
# 显示图片到当前设备
display(cells, method = "raster", all = TRUE)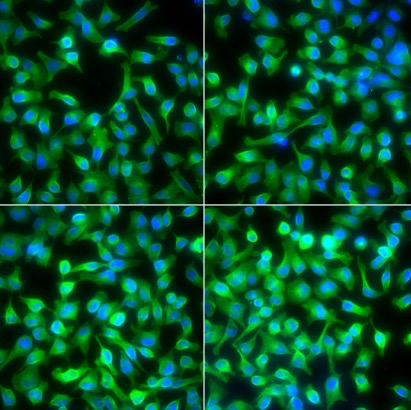
2)二值化处理
# 定义窗口阈值
nmask = thresh(nuc, w=10, h=10, offset=0.05)
# 打开图片
nmask = opening(nmask, makeBrush(5, shape='disc'))
# 对图像进行填充
nmask = fillHull(nmask)
# 执行图像二值化处理
nmask = bwlabel(nmask)
display(nmask, method = "raster", all=TRUE)
3)寻找边界
# 选择cel元素大于0.1的
ctmask = opening(cel>0.1, makeBrush(5, shape='disc'))
# 在图像中找到相邻区域之间的边界
cmask = propagate(cel, seeds=nmask, mask=ctmask)
display(ctmask, method = "raster", all=TRUE)
4)图像组合拼接
# 绘制边界
segmented = paintObjects(cmask, cells, col='#FFFFFF')
# 组合
segmented = paintObjects(nmask, segmented, col='#FFF2CC')
display(segmented, method = "raster", all=TRUE)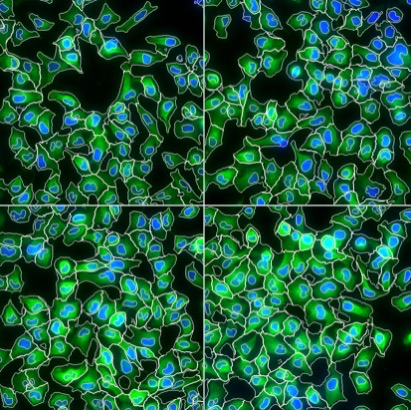
处理起来很方便,相比较PS/AI可能更方便,但是这个需要对图像基础的一些处理方法有一点了解。
参考资料:
1.https://bioconductor.org/packages/release/bioc/vignettes/EBImage/inst/doc/EBImage-introduction.html
 浙公网安备 33010802011761号
浙公网安备 33010802011761号