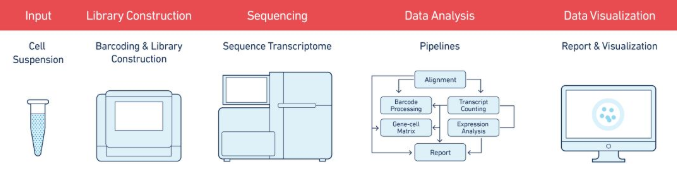
单细胞测序的应用领域非常多,比如CNV检测,ATAC-seq等, 其中引起广泛关注的,就是单细胞在转录组学的应用,即single cell RNA sequencing, 简称scRNA-seq,下面我们从实验设计到数据分析介绍整个单细胞设计流程:
1.实验设计
1)尽可能的避免引起批次效应和混淆生物学因素
2)UMIs标记的使用(定量标准,对照):如使用相对长的UMI等
3)Spike-ins(分子标记,定量标准,对照)使用
4)细胞数量 VS 读取深度的考量

2.原始数据处理、质控
1)Read QC 和 Trimming:常用软件 FASTQC, cutadapt
2)Mapping: 常用的有STAR , Salmon , kallisto
3)定量:i、小的数据集,不含UMIs建议使用 featureCounts ;ii、大的数据集不含UMIs建议 Salmon, kallisto ;iii、UMIs数据集建议 UMI-tools + featureCounts
3.表达矩阵处理
1)Cell QC,使用 scater 等
2)数据标准化使用 scran
3)批次效应处理(如果存在),可以使用如: RUVs 、 mnnCorrect 、 ComBat 等
4.生物学意义探索
1)特征选择, M3Drop
2)聚类和Marker基因鉴定,≤5000 cells : SC3;>5000 cells : Seurat
3)Pseudotime( 拟时序分析:拟时序是一种测量方式,表示的是一个细胞在某种转变过程中的“进展”,进展越少越接近原始细胞状态,进展越多越接近终点细胞状态):i、不同时间点: TSCAN ;ii、小数据集/未知数量的分支: Monocle2 ;iii、大型连续数据集: destiny
4)差异表达分析,此处可选择的工具有很多了,如: edgeR 、 MAST 、 scde 等等。
参考资料:
1.https://scrnaseq-course.cog.sanger.ac.uk/website/index.html
2.https://www.10xgenomics.com/cn/resources/support-documentation/
 浙公网安备 33010802011761号
浙公网安备 33010802011761号