clusterProfiler 是一个非常好用的R包,可以用来做各种富集分析,如GO、KEGG、DO(Disease Ontology analysis)、Reactome pathway analysis以及GSEA富集分析等。而除了富集分析,它还可以非常方便的做蛋白、基因等ID之间的转换。
# 安装R包
if (!requireNamespace("BiocManager", quietly = TRUE))
install.packages("BiocManager")
BiocManager::install("clusterProfiler")
BiocManager::install("org.Hs.eg.db")以人的蛋白、基因名之间的转换为例:
# 加载R包
library(clusterProfiler)
library(org.Hs.eg.db)查看org.Hs.eg.db数据库中支持多少类ID的转换:

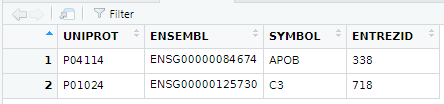
以Uniport ID转换为例
gene <- bitr(
c("P04114", "P01024"),
fromType = "UNIPROT",
toType = c("UNIPROT", "ENSEMBL", "SYMBOL", "ENTREZID"),
OrgDb = org.Hs.eg.db
)
当然,我们也可以尝试其他ID的转换,本文点到为止。
参考文章:
1.http://www.bioconductor.org/packages/release/bioc/vignettes/clusterProfiler/inst/doc/clusterProfiler.html

 浙公网安备 33010802011761号
浙公网安备 33010802011761号
hhhhhh
比较好奇 我该怎么把转换好的ID作为一列加入原来的表达矩阵呢?
陈浩
试试merge
陈浩
bitr用法,直接可以访问:https://yulab-smu.github.io/clusterProfiler-book/chapter14.html#bitr