火山图(Volcano Plot)是大家在文献里面经常看到的图,火山图的横坐标通常用log2(fold change)表示,差异越大的基因分布在两端,纵坐标用-log10(pvalue)表示。
现在高分文章的火山图绘制更是花样多变。以下以ggplot2为绘图方法讲解如何将差异标注加至火山图。
数据结构如下:
R Code如下:
library(airway)
library(magrittr)
library("DESeq2")
# 加载演示数据
data("airway")
airway$dex %<>% relevel("untrt")
# DESeq2计算差异
dds <- DESeqDataSet(airway, design = ~ cell + dex)
dds <- DESeq(dds, betaPrior = FALSE)
res1 <- results(dds, contrast = c("dex", "trt", "untrt"))
res1 <-
lfcShrink(dds,
contrast = c("dex", "trt", "untrt"),
res = res1)
res2 <- results(dds, contrast = c("cell", "N061011", "N61311"))
res2 <-
lfcShrink(dds,
contrast = c("cell", "N061011", "N61311"),
res = res2)
res3 <- as.data.frame(res2)
# 得到所有差异矩阵
Data <- res3
Data <- Data[!is.na(Data$padj),]
# 计算上下调关系
Data$threshold = factor(ifelse(
Data$padj < 0.05 &
abs(Data$log2FoldChange) >= 1,
ifelse(Data$log2FoldChange >= 1 , 'Up', 'Down'),
'-'
),
levels = c('Up', 'Down', '-'))
Data$Gene <- rownames(Data)
# ggplot2 画火山图
ggplot(Data, aes(
x = log2FoldChange,
y = -log10(padj),
color = threshold
)) +
geom_point(alpha=0.5,size=3,shape=19, fill='white') +
scale_color_manual(values = c("#DC143C", "#00008B", "#808080")) + #确定点的颜色
geom_text_repel(
# 添加差异标签
data = Data[Data$padj < 0.01 & abs(Data$log2FoldChange) > 2,],
aes(label = Gene),
size = 3,
segment.color = "black",
show.legend = FALSE
) + #添加关注的点的基因名
theme_bw() + #修改图片背景
theme(legend.title = element_blank()) + #不显示图例标题
xlim(-7.5, 7.5) +
ylab('-log10 (P.adjusted Value)') + #修改y轴名称
xlab('log2 (FoldChange)') + #修改x轴名称
geom_vline(
xintercept = c(-1, 1),
lty = 3,
col = "black",
lwd = 0.5
) + #添加横线|FoldChange|>2
geom_hline(
yintercept = -log10(0.05),
lty = 3,
col = "black",
lwd = 0.5
)#添加竖线P.Value<0.05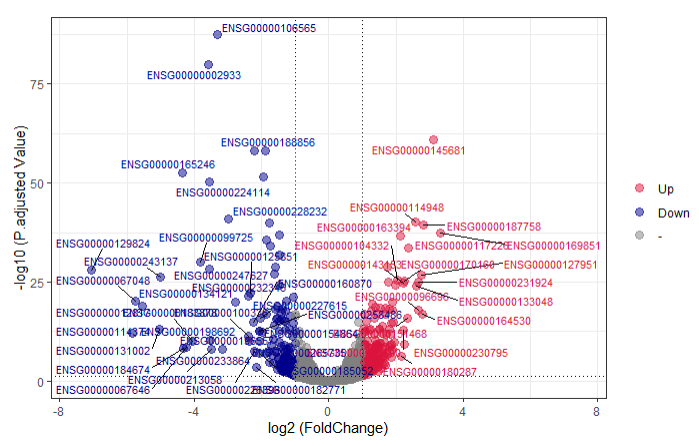
以上就是一个简单的火山图绘制完整代码。

 浙公网安备 33010802011761号
浙公网安备 33010802011761号
陈浩
看到很多朋友问怎么用ggplot2在火山图上怎么显示某一个或者某几个基因,可以利用geom_text_repel来完成,输入的数据框和需要显示的基因的fc和p值对应(即x轴和y轴),如下:
geom_text_repel(
# 添加差异标签
data = Data[Data$P.Value < 0.05 & abs(Data$logFC) > 1,],
aes(label = Gene),
size = 3,
segment.color = “black”,
show.legend = FALSE
)
此处数据框也可以自定义需要显示的点的名称,比如某一个或者某几个。