一般我们做GO注释要用到 clusterProfiler 的enrichGO函数。我们先看看函数的调用参数:

在这里面我们要用到OrgDb的数据库,很多时候我们用的数据库在bioconductor中直接下载。
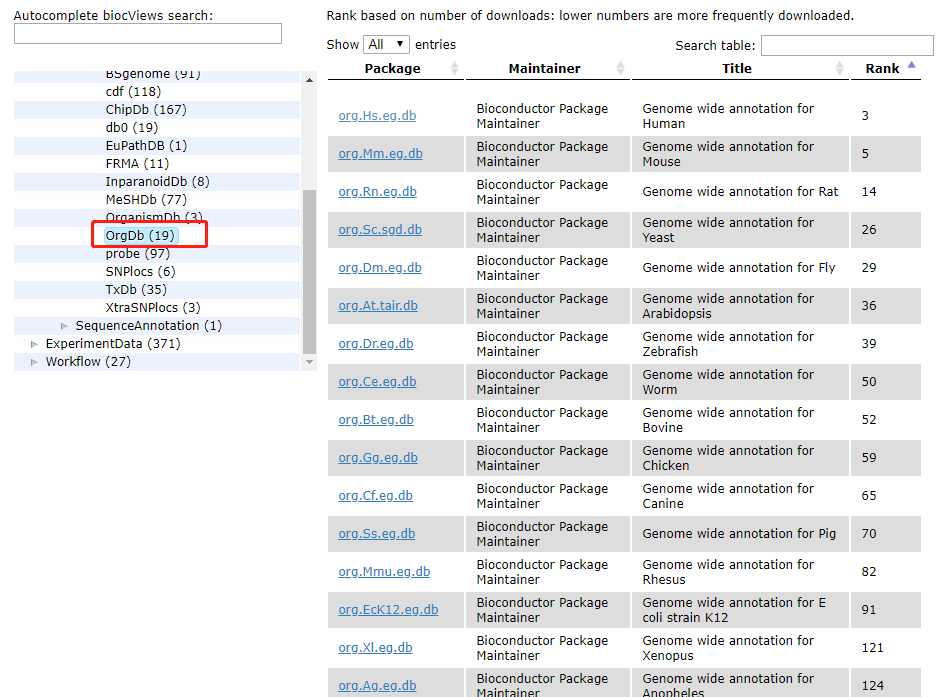
但是一部分非模式生物就不再我们的Bioconductor中,这里的难点不是说不会用数据库,而是不知道如何去下载?或者说去哪里下载?这里其实在Y叔的博客和一些生信的公众号也多有介绍,这里就简单说说吧,更多细节可以查看参考文章。
这里我们主要利用 AnnotationHub 包去检索,然后生成数据库对象供 clusterProfiler 调用。
下面一个简单的例子:
library(AnnotationHub)
library(biomaRt)
library(clusterProfiler)
hub <- AnnotationHub()
query(hub,"Glycine max")
annoDB <- hub[["AH66196"]]
columns(annoDB)
id <- read.table("id.txt") #gene id
colnames(id) <- "id"
id <- as.character(id$id)
go <- enrichGO(id, OrgDb = annoDB, pvalueCutoff=1, qvalueCutoff=1, readable = T)
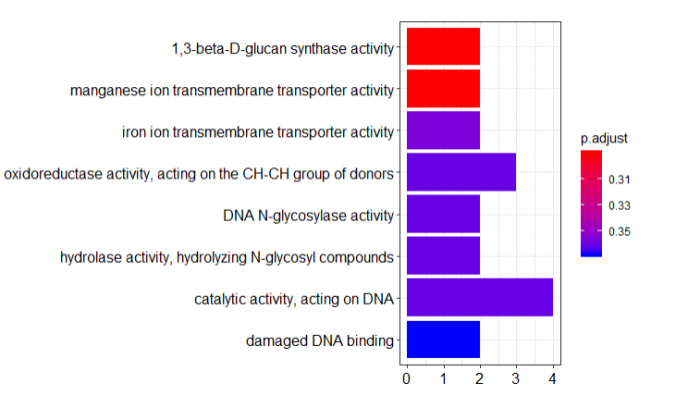
barplot(go)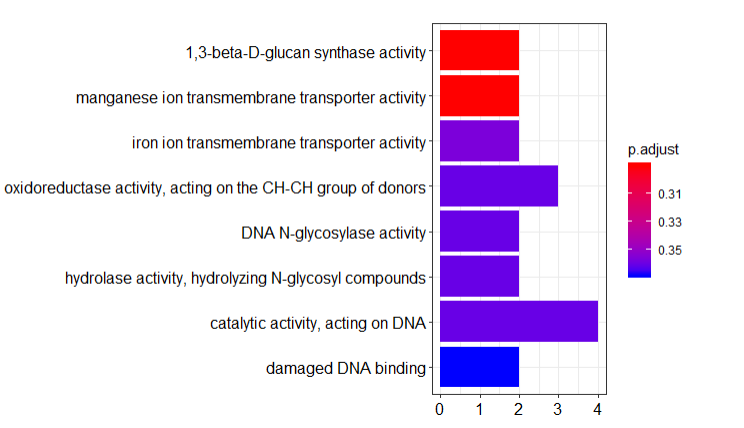
最后就生成了我们久违的GO分析分析的barplot。
好奇的我去看了下发生了什么事情,看看再 AnnotationHub 中发生里什么事情。首先看了下 AnnotationHub 对象
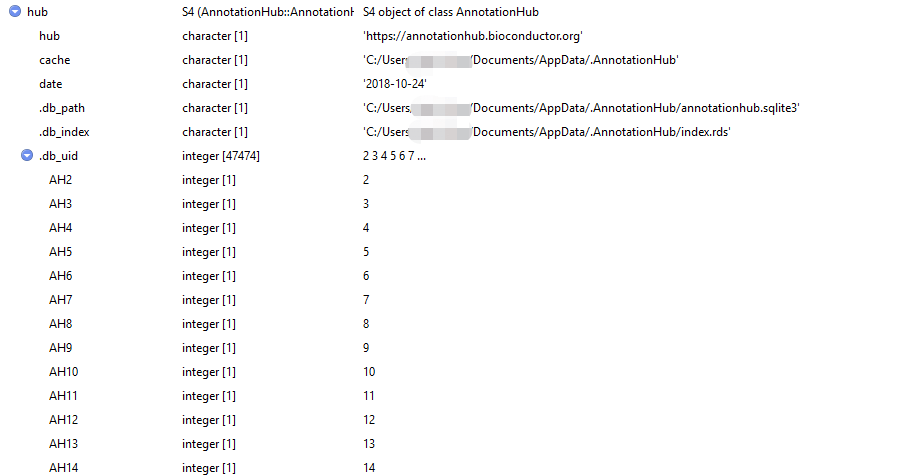
原来是检索了 AnnotationHub 的数据库,那些不再Bioconductor的数据库中的物种原来在这里。
最后打开这个sqlite看了下内容:
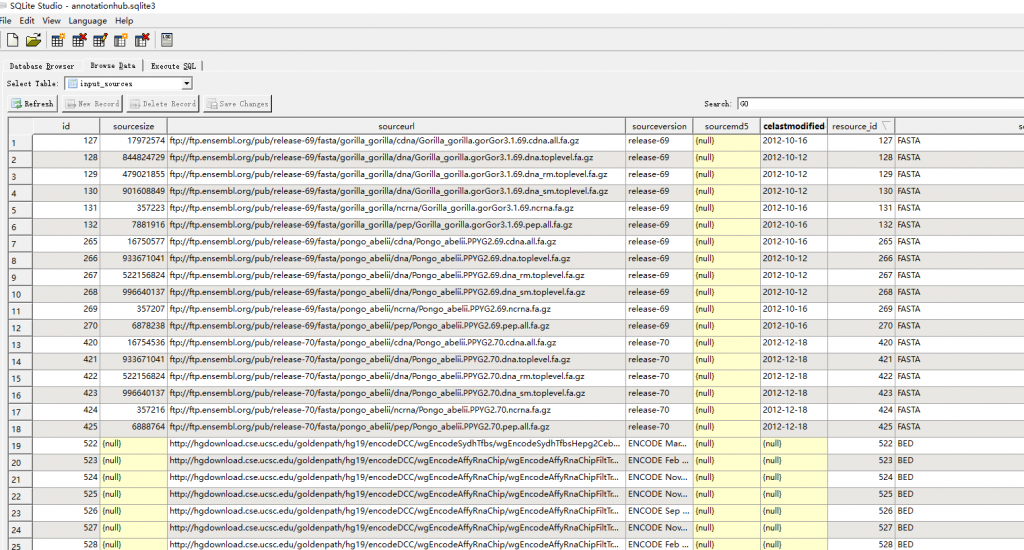
几乎所有的注释信息都在这个数据库中,所以当大家遇见一些在Bioconductor中无法找到的注释信息不妨去看看这个。
这个数据库链接上传一份备用:
一点小小的总结分享给大家。
参考文章:
1.http://bioconductor.org/packages/release/bioc/vignettes/clusterProfiler/inst/doc/clusterProfiler.html
2.https://guangchuangyu.github.io/cn/2017/07/clusterprofiler-maize/
3.https://www.jianshu.com/p/9c9e97167377
 浙公网安备 33010802011761号
浙公网安备 33010802011761号