SWATH MS技术的一大难题就是对复杂的质谱数据的解析。这是因为在SWAHT采集模式下,一个窗口中的所有母离子的全部碎片离子都呈现在了一张谱图中,使用传统的蛋白质质鉴定软件不能有效解析这些谱图。
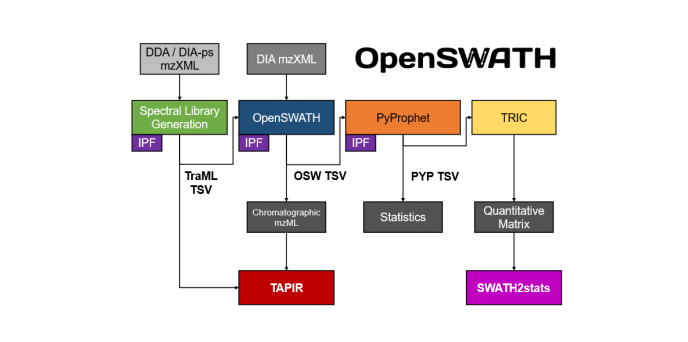
The OpenSWATH Workflow enables targeted data analysis of data-independent acquisition (DIA) or SWATH-MS proteomic data. The main workflow consists of OpenSWATH, PyProphet, TRIC, IPF and TAPIR. This website provides documentation on installation and application of the tools.
http://www.openswath.org
OpenSWATH将SWATH数据的保留时间、碎片离子峰的形状、碎片离子的强度等信息分别与目标谱图库的对应信息做比较并打分(subscore),再使用半监督学习算法对每一个目标肽的所有子离子峰(trace)进行迭代训练、分类,然后线性合并这些subscore,得到一个总得分值。 为了评价结果的可靠性,该算法使用了一种类似传统蛋白质数据库检索的decoy思想。
OpenSWATH安装下载方式:
Docker版:
docker pull openswath/openswath:latest注意:使用docker版需要安装Dokcer。请访问:https://www.docker.com
Windows版:
请访问:https://abibuilder.informatik.uni-tuebingen.de/archive/openms/OpenMSInstaller/release/latest/
Linux&MAC版:
请访问:https://abibuilder.informatik.uni-tuebingen.de/archive/openms/OpenMSInstaller/release/latest/
对应下载安装即可使用。
一个示例(docker版):
step1.打开docker
docker run --name osw_tutorial --rm -v ~/Desktop/:/data -i -t openswath/openswath:lateststep2.运行帮助命令
# Execute OpenSwathWorkflow in docker
OpenSwathWorkflow --help
# Execute PyProphet in docker
pyprophet --help
# Execute TRIC in docker
feature_alignment.py --help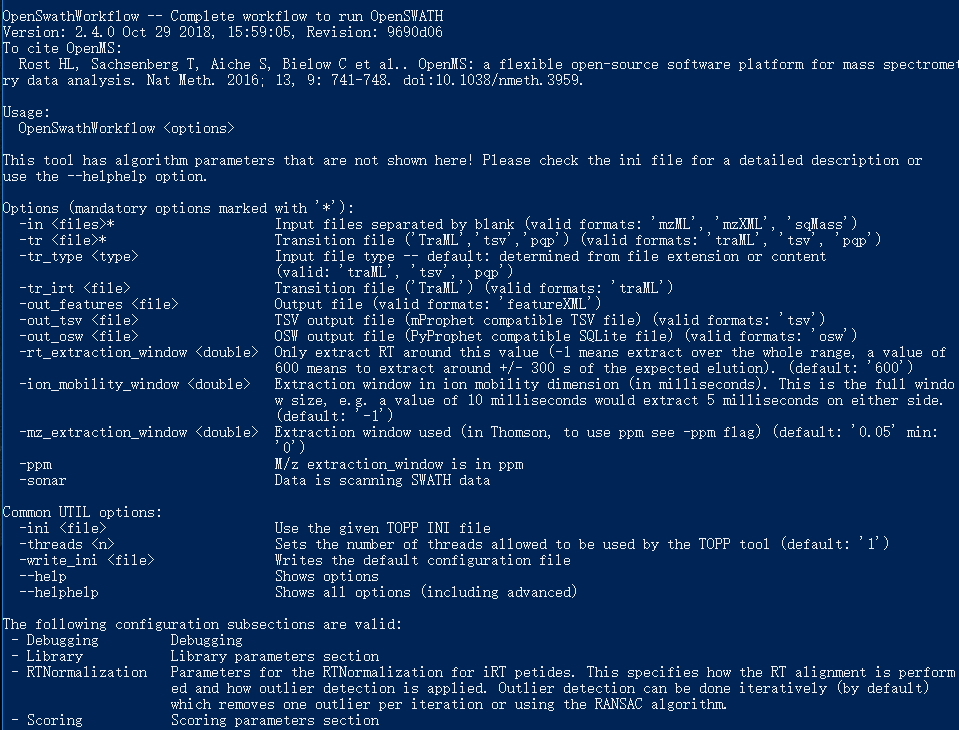
step3.一段示例命令
OpenSwathWorkflow \
-in /data/data.mzML
-tr /data/library.tsv \
-tr_irt /data/iRT_assays.TraML \
-swath_windows_file /data/SWATHwindows_analysis.tsv \
-sort_swath_maps -batchSize 1000 \
-readOptions cacheWorkingInMemory -tempDirectory /tmp/ \
-use_ms1_traces \
-mz_extraction_window 50 -ppm \
-mz_correction_function quadratic_regression_delta_ppm \
-TransitionGroupPicker:background_subtraction original \
-RTNormalization:alignmentMethod linear \
-Scoring:stop_report_after_feature 5 \
-out_tsv /data/osw_output.tsv \本次简单介绍了OpenSWATH及其安装使用。更多信息请访问参考信息。
参考资料:
1.http://www.openswath.org/en/latest/docs/docker.html
2.https://www.openms.de
3.https://en.wikipedia.org/wiki/OpenMS
 浙公网安备 33010802011761号
浙公网安备 33010802011761号