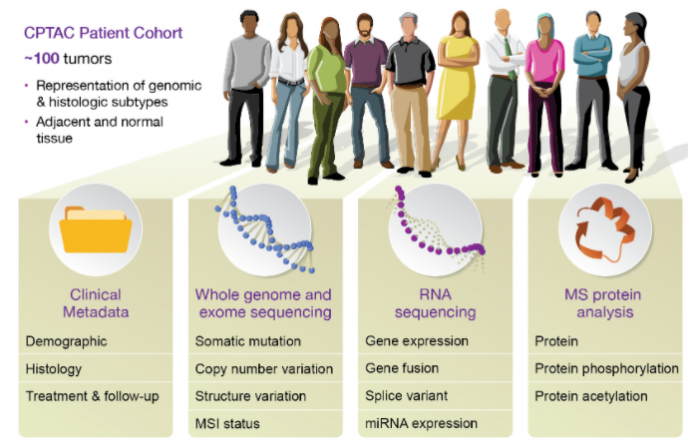
美国国家癌症研究所的临床蛋白质组肿瘤分析联盟 (CPTAC,clinical proteomic tumor analysis consortium) 是一项旨在通过应用大规模蛋白质组和基因组分析或蛋白质组学来加速了解癌症的研究项目。CPTAC 从临床队列中生成全面的蛋白质组学和基因组学数据。
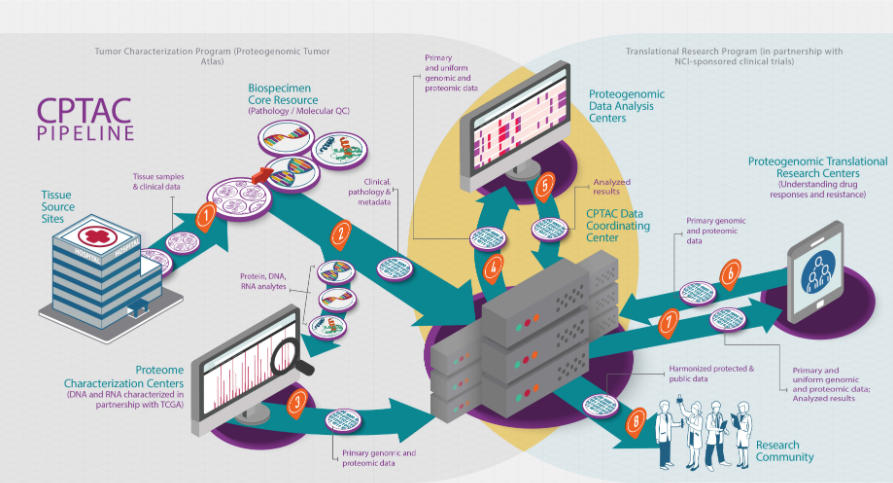
CPTAC数据库用到的蛋白质定量技术主要是基于质谱的检测技术,包括iTRAQ(Isobaric tags for relative and absolute quantification)和TMT(Tandem Mass Tags)。iTRAQ是由美国ABI研发的一种体外同种同位素标记的相对与绝对定量技术,TMT则是由Thermo研发的多肽体外标记技术。两种技术都是采用4种或8种、11种同位素编码的标签,其中最新的TMT技术可以达到16种同位素编码的标签,通过特异性标记蛋白多肽N末端或赖氨酸侧链基团,而后进行串联质谱分析,可同时比较多个不同样品中蛋白质的相对含量。
目前数据集信息如下:
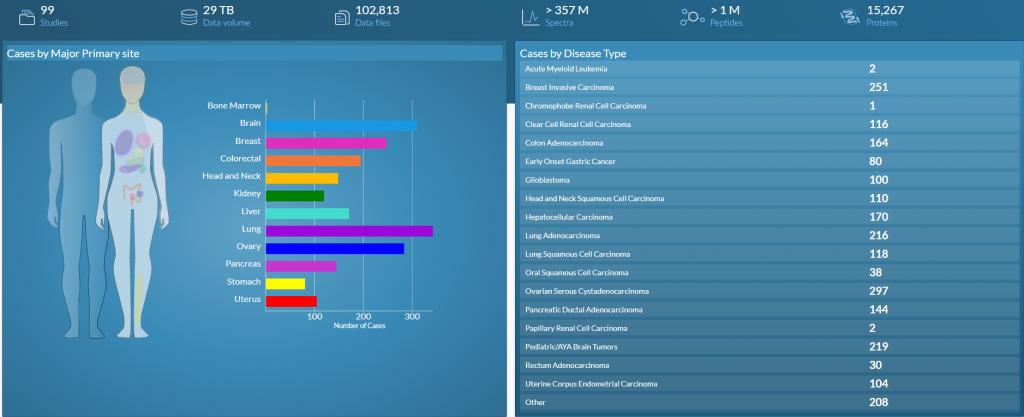
数据访问:https://proteomic.datacommons.cancer.gov/pdc/
例如我们需要研究乳腺癌
https://pdc.cancer.gov/pdc/browse/filters/primary_site:Breast
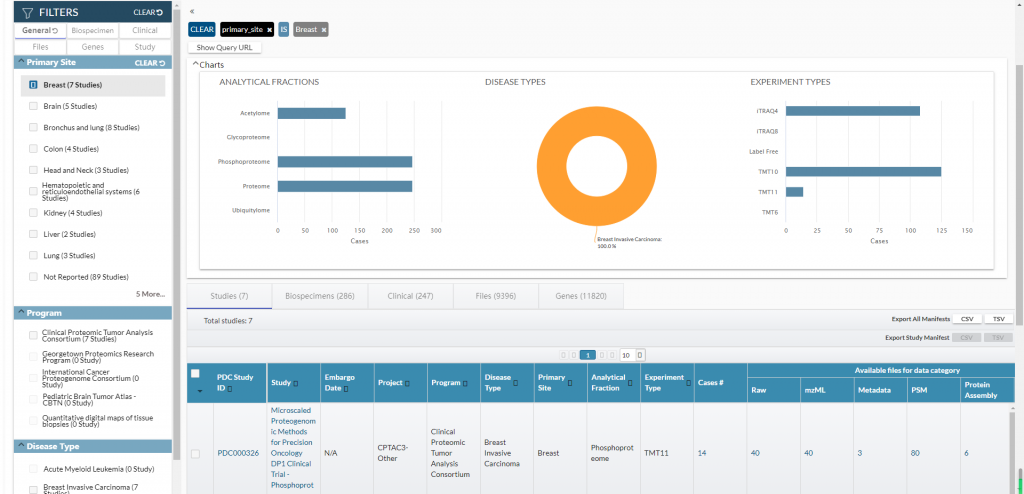
包含了完善的统计信息和数据集信息,点击Study ID查看详细信息
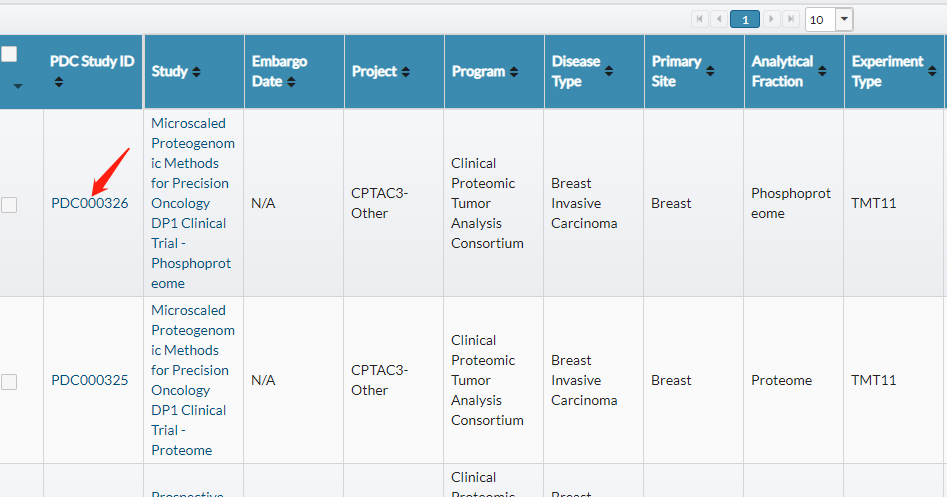
我们可以看到每个研究项目的详细信息:
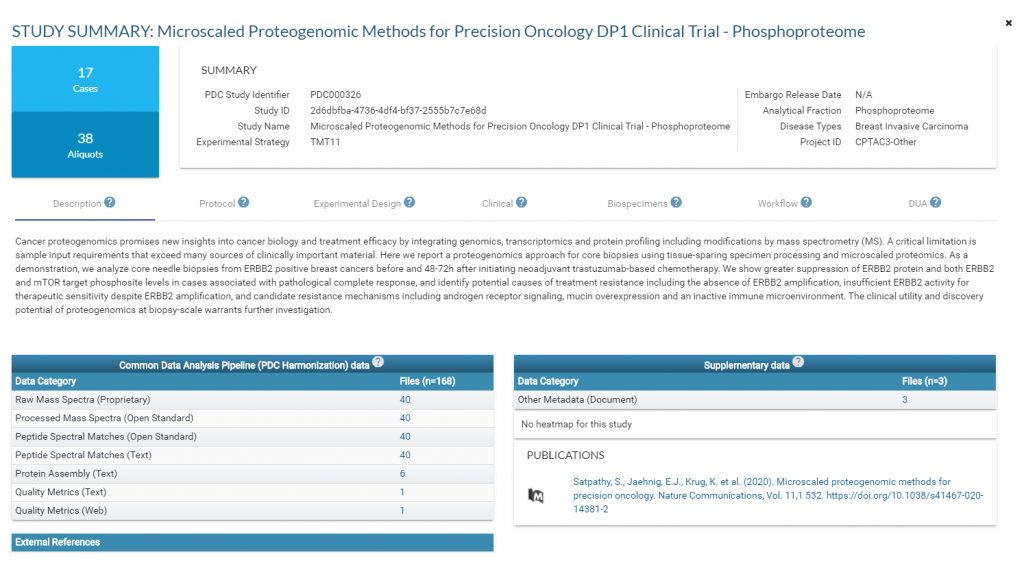
如果我们需要下载原始数据信息点击Files下面的数字
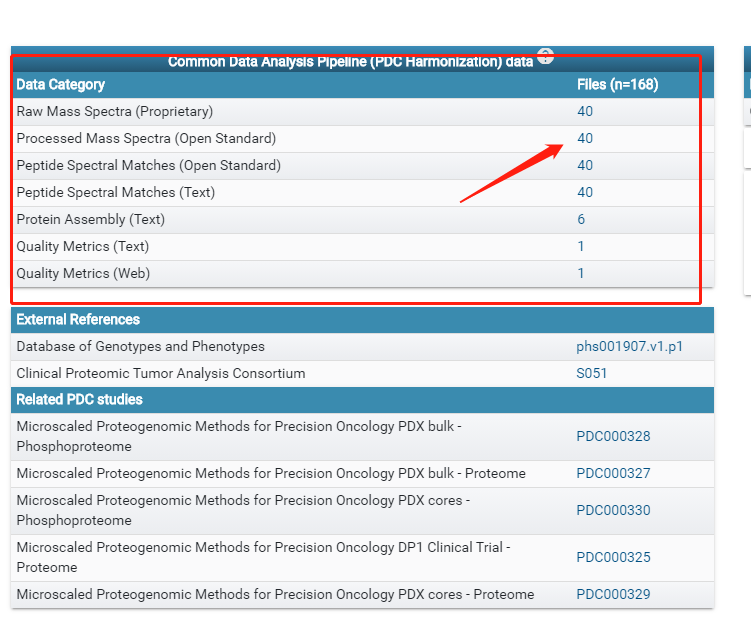
然后点击弹窗的Download下载数据:
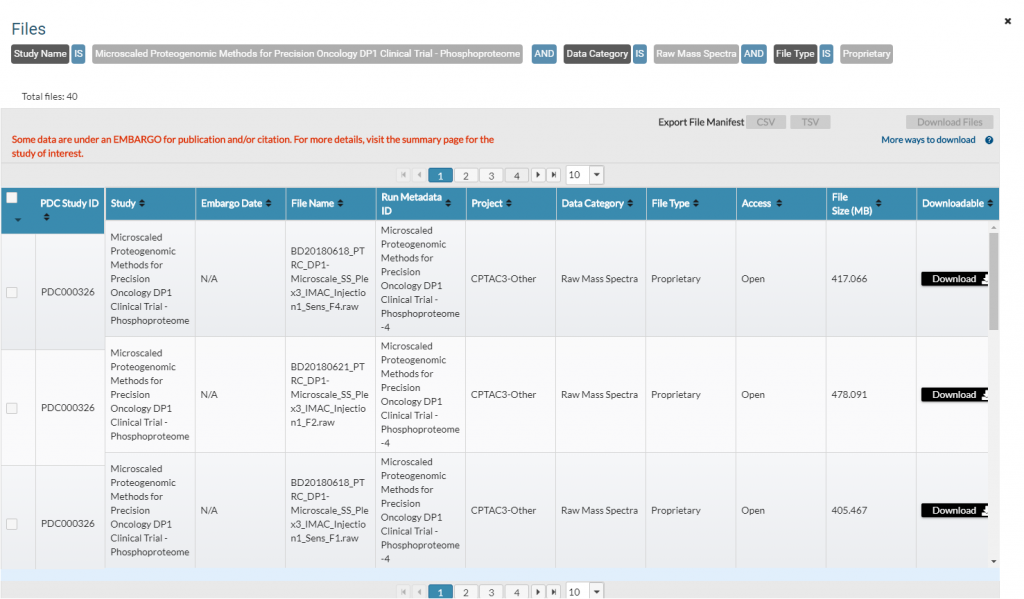
批量下载数据请参考:https://pdc.cancer.gov/pdc/faq/Multiple_Files
当然我们也可以通过CPTAC提供的数据来对数据进行再次分析
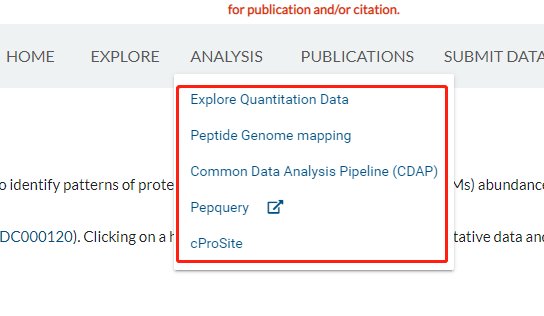
我们也可以根据CDAP来查看和学习蛋白数据的分析流程:
DDA数据分析:
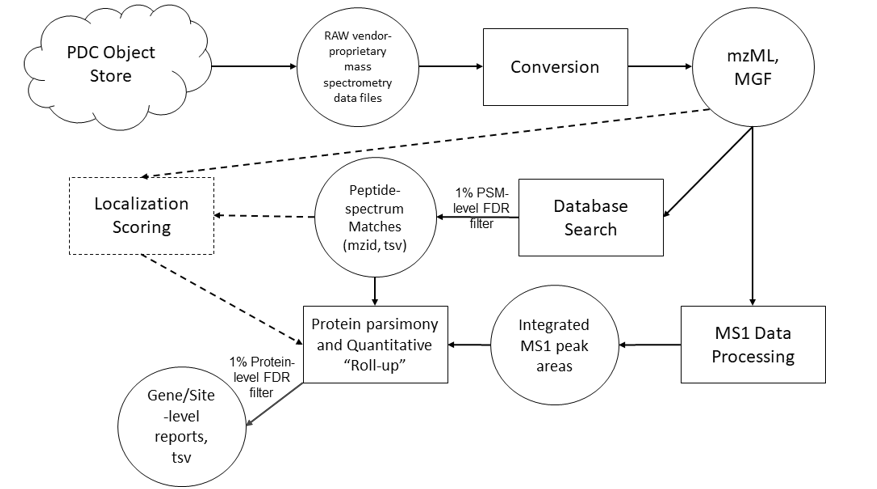
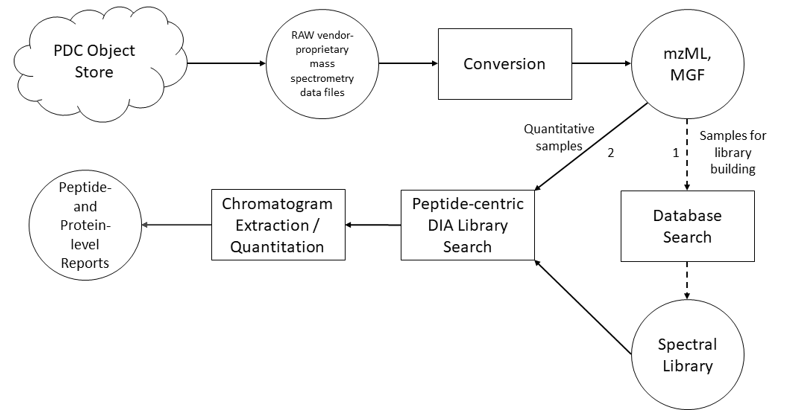
DIA数据分析:
同时也有很多不错的教程讲解CPTAC的挖掘,大家可以学习参考:
教程
- Tutorial 1: CPTAC data introduction
- Tutorial 2: Using pandas to work with cptac dataframes
- Tutorial 3: Joining dataframes with cptac
- Tutorial 4: Understanding multi-indexes
- Tutorial 5: How to keep up to date with new package and data releases
- Tutorial 6: Easy integration with R
案例
- Use Case 1: Comparing transcriptomics and proteomics
- Use Case 2: Correlation between clinical attributes
- Use Case 3: Associating clinical variables with omics data
- Use Case 4: How Do Mutations Affect Protein Abundance?
- Use Case 5: Gene Set Enrichment Analysis
- Use Case 6: Comparing Derived Molecular Data with Proteomics
- Use Case 7: Trans Genetics Effects
- Use Case 8: Outliers
- Use Case 9: Clinical Outcomes
- Use Case 10: Pathway diagram overlay
参考文献:
1.https://paynelab.github.io/cptac/tutorial01_data_intro.html
2.https://proteomics.cancer.gov/programs/cptac
 浙公网安备 33010802011761号
浙公网安备 33010802011761号
猜猜
看了陈老师的分享,受益匪浅
某研究生
首先,感谢作者的分享,讲述的也非常详细,就是有一个小小问题,CPTAC的使用介绍里,最后那里的“载此分析”是什么意思呢?是写错了吗?还是说我没有理解(冒昧问一下)
陈浩
感谢指出,这是通(错)假(别)字,通“再次分析”。