全基因组筛选目标是产生并筛选突变细胞的群体,以鉴定出与特定表型相关的基因。现在流行的全基因组CRISPR文库筛选技术被广泛应用于肿瘤及病毒相关调控基因的筛选上。
CRISPR/Cas9的主要优势在于它的简单和通用。CRISPR/Cas9系统有两部分组成:非特异性的核酸内切酶(Cas9)和单链的向导RNA(sgRNA)
整个流程通过同时对sgRNA文库同时敲除全基因组的不同基因,经流式或药物筛选目的细胞表型,并扩增sgRNA区域序列及NGS测序,由此获得显著富集或减少的sgRNA所对应的基因。
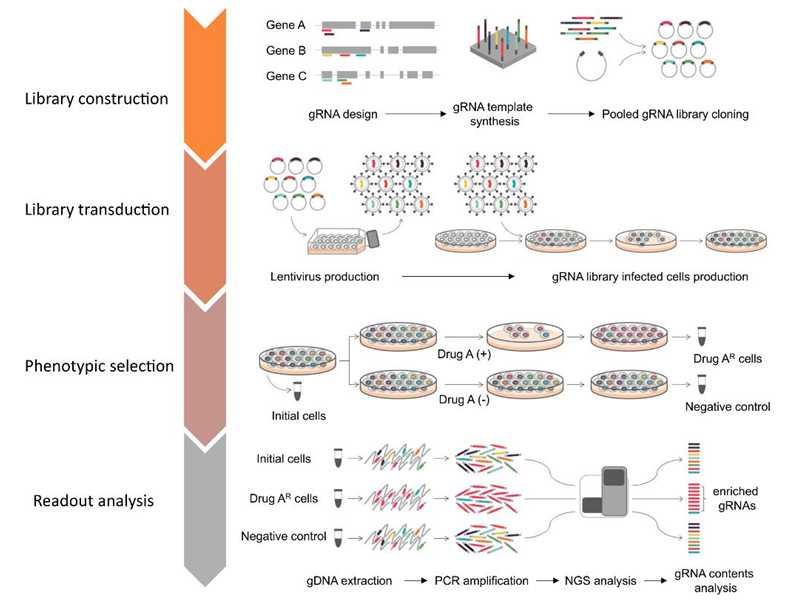
其下游数据分析可用的方法也比较多,相对来说MAGeCK目前来说是比较方便的分析CRISPR/Case9 NGS测序数据的套件,包含了从原始数据到可视化的全流程解决方案。
1)安装
基于conda:
conda install -c bioconda mageck
# 或者建立专用的conda环境
conda create -c bioconda -n mageckenv mageck
source activate mageckenv基于docker:
# 下载镜像
docker pull davidliwei/mageck
# 进入docker环境
docker run -it --volume=`pwd`:/work --workdir="/work" davidliwei/mageck2)数据分析

从下机原始数据开始分析(支持双端测序):
# 生成Conut矩阵
mageck count -l library.txt -n demo --sample-label L1,CTRL --fastq test1.fastq test2.fastq
# 对于双端测序
mageck count -l library.txt -n demo --sample-label L1,CTRL --fastq test1_1.fastq test2_1.fastq --fastq-2 test1_2.fastq test2_2.fastq
# 差异检测寻找靶基因
mageck test -k demo.count.txt -t L1 -c CTRL -n demo其中library文件格式如下(支持txt和csv两种)

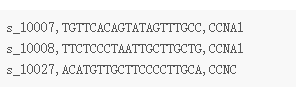
3)数据可视化-MAGeCKFlute
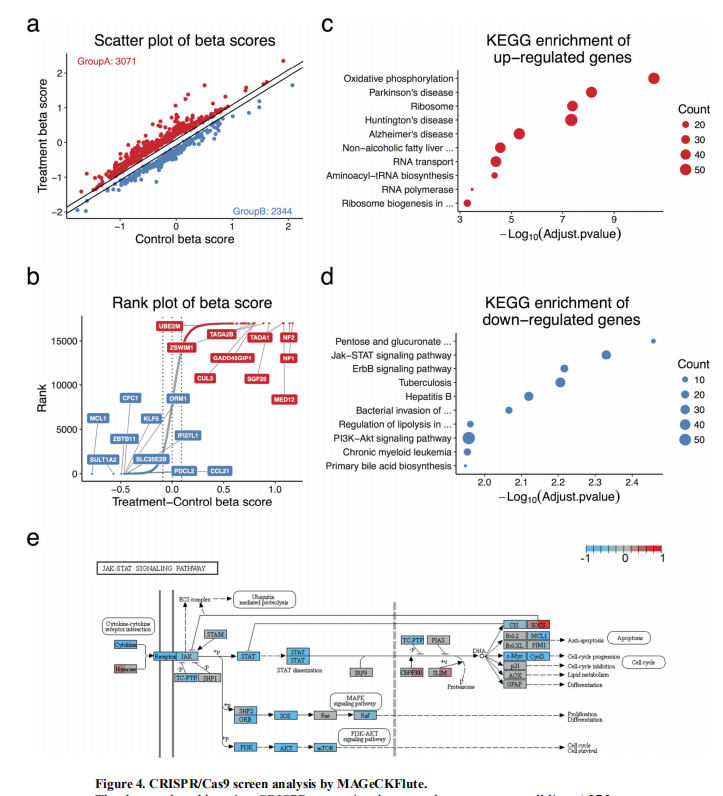
MAGeCKFlute 是和 mageck 配套的下游数据可视化的R包,非常方便。
# 安装
if(!"MAGeCKFlute" %in% installed.packages()) BiocManager::install("MAGeCKFlute")
if(!"clusterProfiler" %in% installed.packages()) BiocManager::install("clusterProfiler")
if(!"ggplot2" %in% installed.packages()) BiocManager::install("ggplot2")
library(MAGeCKFlute)
library(clusterProfiler)
library(ggplot2)用上一步产生的结果文件sgRNA和基因summary文件:count_summary.txt、gene_summary.txt、sgrna_summary.txt等几个文件作为 MAGeCKFlute 输入,后续的操作非常简单了,都是封装好的函数,直接对照文档使用即可。
参考资料:
1.https://hpc.nih.gov/apps/MAGeCK.html
2.https://sourceforge.net/p/mageck/wiki/Home/
3.https://bitbucket.org/liulab/mageck/src/master/
4.https://bioconductor.org/packages/devel/bioc/vignettes/MAGeCKFlute/inst/doc/MAGeCKFlute.html

 浙公网安备 33010802011761号
浙公网安备 33010802011761号