g:Profiler是继DAVID后另一款功能强大的在线富集分析和基因ID转换工具。

其主要功能包含四大块:
- g:GOSt:功能富集分析工具
- g:Convert:基因ID、蛋白名、转录本等ID相互转换
- g:Orth:跨物种同源性分析
- g:SNPense:定位SNP的rs识别染色体位置,基因信息和变异类型
1) g:GOSt
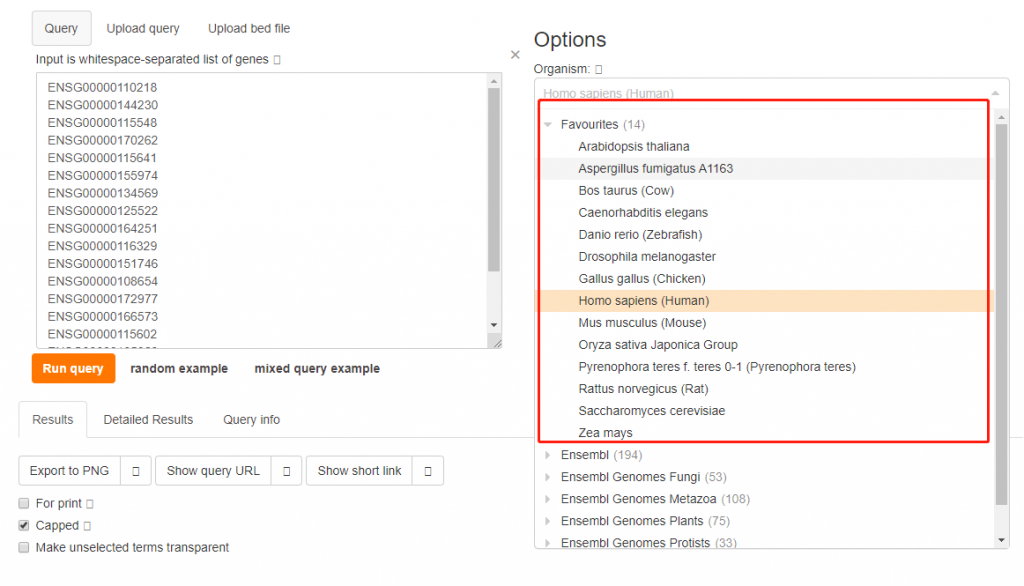
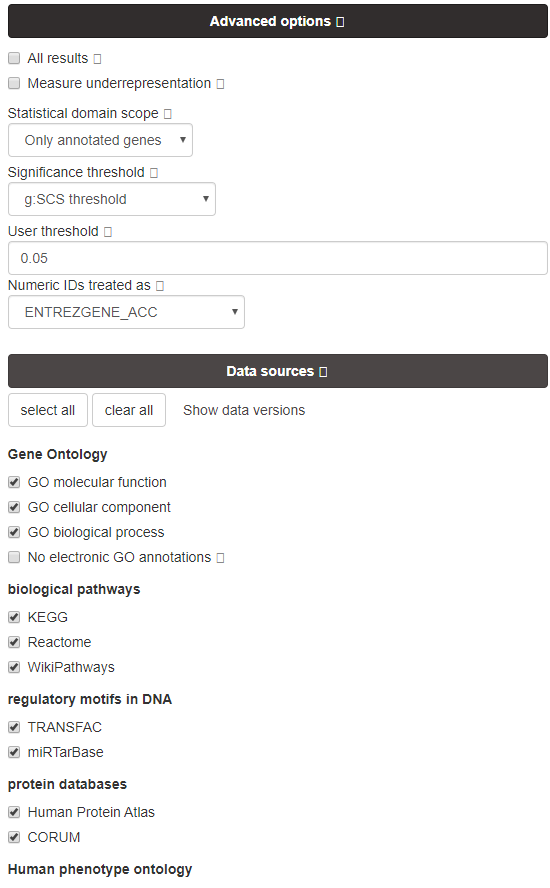
包含了常见的物种,高级参数中包含了选择注释的数据库信息默认是全部选择,我们输入基因信息,点击 Run query 输入富集结果:每一种颜色代表不同的功能或者数据库,点的大小代表该通路中含有的总基因的数量(非提交的基因数)

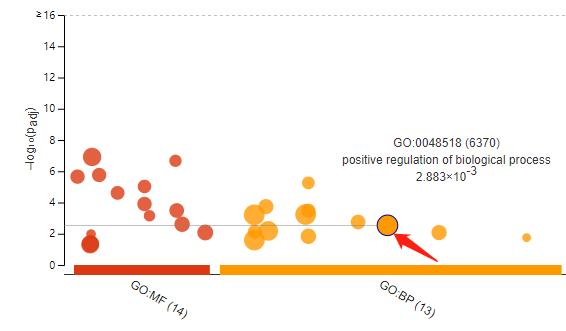
支持鼠标点选和导出图片,使用很方便,我们也可以点击 Detailed Results 查看注释细节信息:
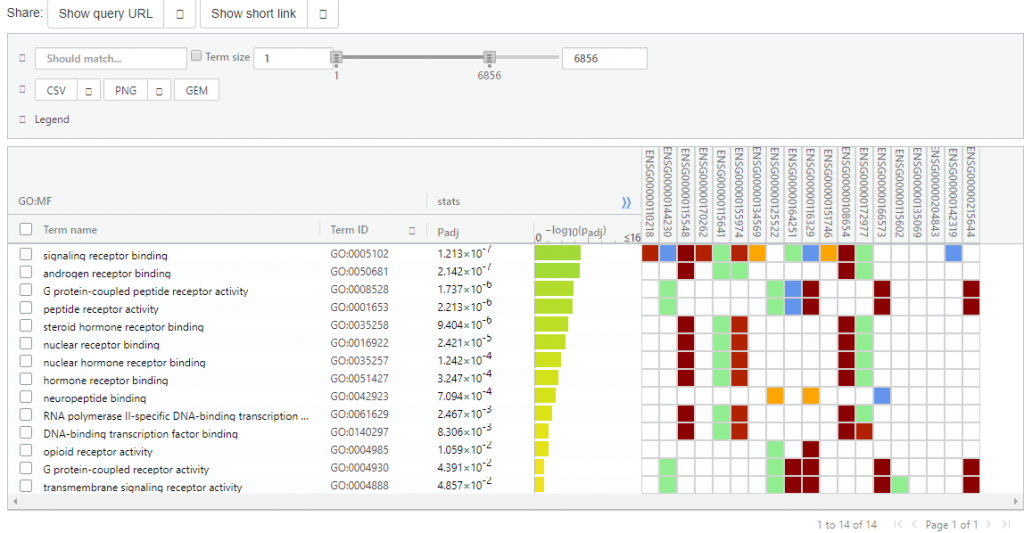
每种颜色代表不同的类型,如下:

2) g:Convert
此功能比较常用,也比较简单。输入列表后,选择物种,然后选择需要转换的目标类型和你输入的列表类型即可完成快速转换。

3) g:Orth
结果中展示initialENSG和 ortholog ENSG 的同源信息

4) g:SNPense
这个功能输入也相对简单,输入snp位点信息,mappingrs位点的染色体、基因等信息
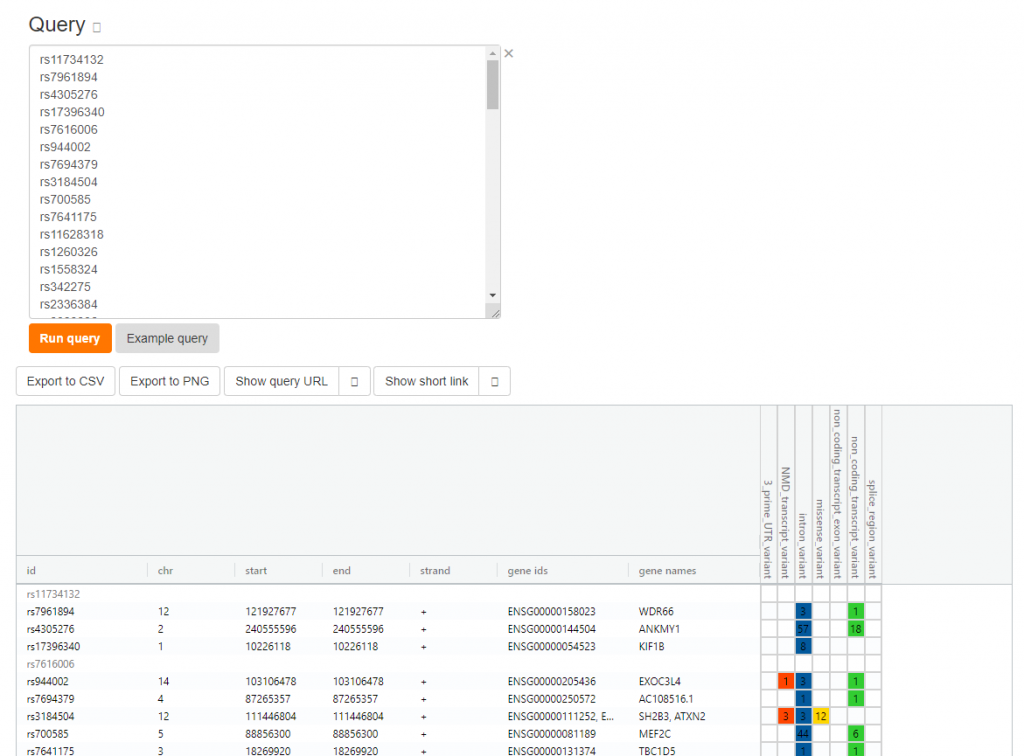
总体来说,g:Profiler的易用性和可视化做的非常棒,科研中不可或缺的工具。
同时官方也设计了API,支持从R之间调用:
# 安装
install.packages("gprofiler2")# 一个示例
library(gprofiler2)
gostres <- gost(query = c("X:1000:1000000", "rs17396340", "GO:0005005", "ENSG00000156103", "NLRP1"),
organism = "hsapiens")
# 绘图
gostplot(gostres, capped = FALSE, interactive = FALSE)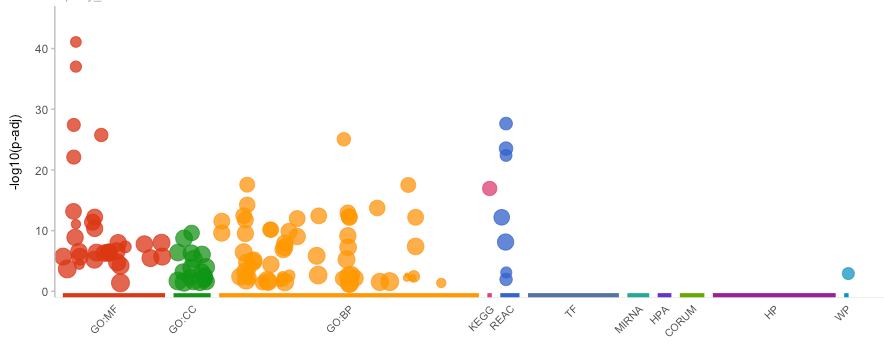
参考资料:
1.http://biit.cs.ut.ee/gprofiler/gost
2.https://cran.r-project.org/web/packages/gprofiler2/vignettes/gprofiler2.html
 浙公网安备 33010802011761号
浙公网安备 33010802011761号