UCSC Genome Browser基因组浏览器在大规模高通量数据的可视化和比较分析研究中发挥着重要的作用,关于其介绍更是不胜枚举,本文主要介绍其bigwig文件在基因组浏览器中的可视化操作。
bigWig 格式对于将在基因组浏览器中显示密集、连续数据非常有用,后缀名:.bw和.bigwig,其数据必须是连续的并且由大小相等的元素组成。
bigWig 文件可以从wig文件(http://www.genome.ucsc.edu/goldenPath/help/wiggle.html)或者bedgraph文件(http://www.genome.ucsc.edu/goldenPath/help/bedgraph.html)转换生成。
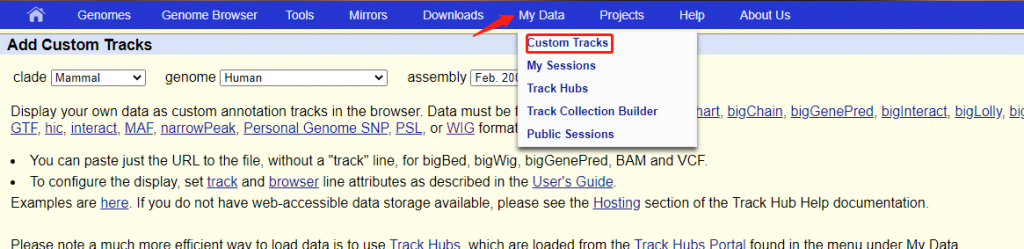
1)当我们准备好 .bw和.bigwig 文件后,打开主页:http://www.genome.ucsc.edu/cgi-bin/hgCustom,选择Custom Tracks
2)我们在输入框提交如下信息:
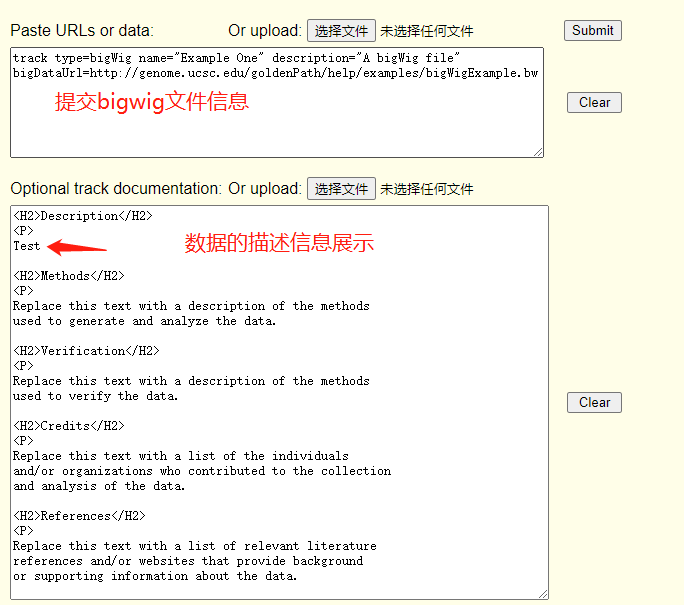
点击submit提交数据。
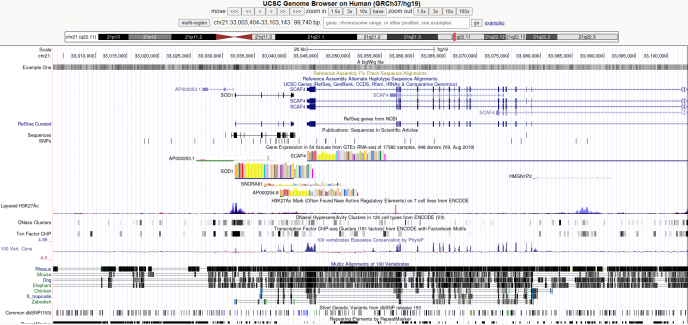
3)可视化数据

如果我们还要添加多条tracks点击下方add custom tracks即可,此处演示我们点击提交(go),如下:

然后我们输入位置信息查看我们的track:

点击我们的数据,会跳转到我们刚定制的描述页面:

如果要分享你的track,可以通过http://www.genome.ucsc.edu/cgi-bin/hgSession创建账号,然后来分享你的track。

参考资料:
1.http://genome.ucsc.edu/FAQ/FAQformat.html#format6
2.http://www.genome.ucsc.edu/goldenPath/help/bigWig.html
3.http://www.genome.ucsc.edu/index.html
 浙公网安备 33010802011761号
浙公网安备 33010802011761号
hcj
如果是在本地下载了ucsc,也可以用上述方法添加track吗
陈浩
本文介绍的方式是online的形式,如果你需要搭建本地版的USCS可以参考:https://genome-euro.ucsc.edu/goldenPath/help/gbic.html,添加track的方式是一样的,但是无法分享