细胞间的通讯关系的分析方法有很多,诸如SingleCellSignalR,iTALK, CellPhoneDB 和NicheNet等,他们主要使用一个配体/受体基因对来进行分析或者未考虑其他重要的信号辅助因子等。
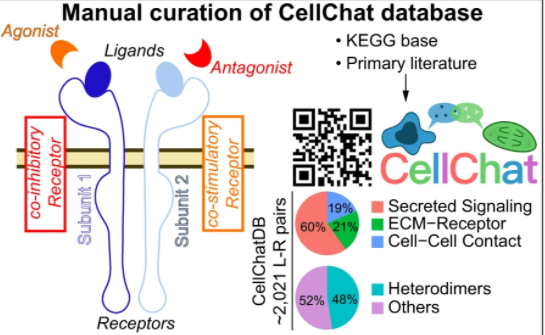
CellChat是一个开源R包,主要用于细胞间通讯的分析和可视化 。作者首先建立了一个信号分子相互作用的数据库,充分考虑了配体-受体相互作用的已知结构组成;然后,CellChat使用质量作用模型,以及对细胞组的差异表达分析和统计测试,在给定的scRNA-seq数据内推断细胞状态特定的信号通讯。
CellChat同时还提供了多个可视化输出,并通过网络分析工具,模式识别方法和多种学习方法对细胞间通讯进行定量表征和比较。
CellChat将单细胞表达数据作为输入,并结合配受体及其辅助因子的相互作用来模拟细胞间通讯:
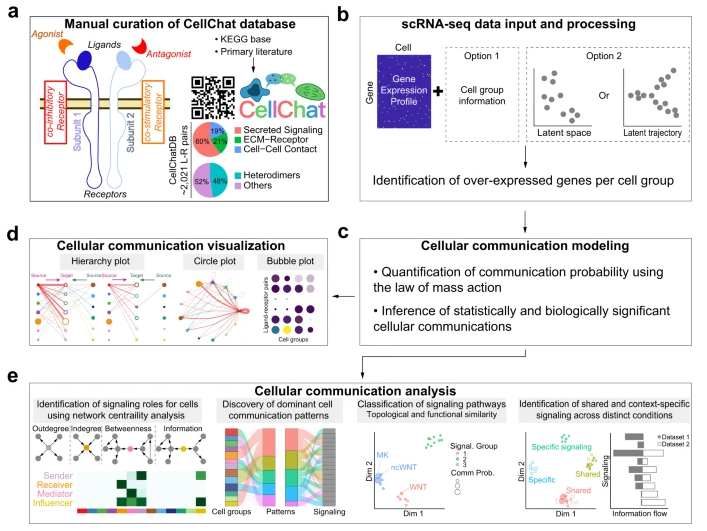
CellChat可以识别通讯模式并预测很少研究的通路的功能,CellChat也能够量化所有重要信号通路之间的相似性,根据细胞通讯网络的相似性对其进行分组分析。

CellChat揭示连续细胞谱系相关的信号事件

CellChat还有很多功能分析,如:预测空间共定位细胞群之间的关键信号事件、同一组织中不同时间的细胞间通讯情况、不同状态之间的保守和状态特异的通信模式等,具体可以参考作者的文章。
安装 CellChat :
# 注意需要先安装如下依赖
# NMF (>= 0.23.0) install.packages('NMF')
# circlize (>= 0.4.12) devtools::install_github("jokergoo/circlize")
# Install ComplexHeatmap devtools::install_github("jokergoo/ComplexHeatmap")
devtools::install_github("sqjin/CellChat")官方教程(很详细):
- Full tutorial for CellChat analysis of a single dataset with detailed explanation of each function
- Full tutorial for comparison analysis of multiple datasets
- Comparison analysis of multiple datasets with different cellular compositions
- Interface with other single-cell analysis toolkits (e.g., Seurat, SingleCellExperiment, Scanpy)
- Tutorial for updating ligand-receptor database CellChatDB
同时作者也提供了基于shiny的 CellChat 应用(https://github.com/sqjin/CellChatShiny),真可谓是周到,只需要将 CellChat 对象保存cellchat.rds放到shiny应用文件夹下就可以了。
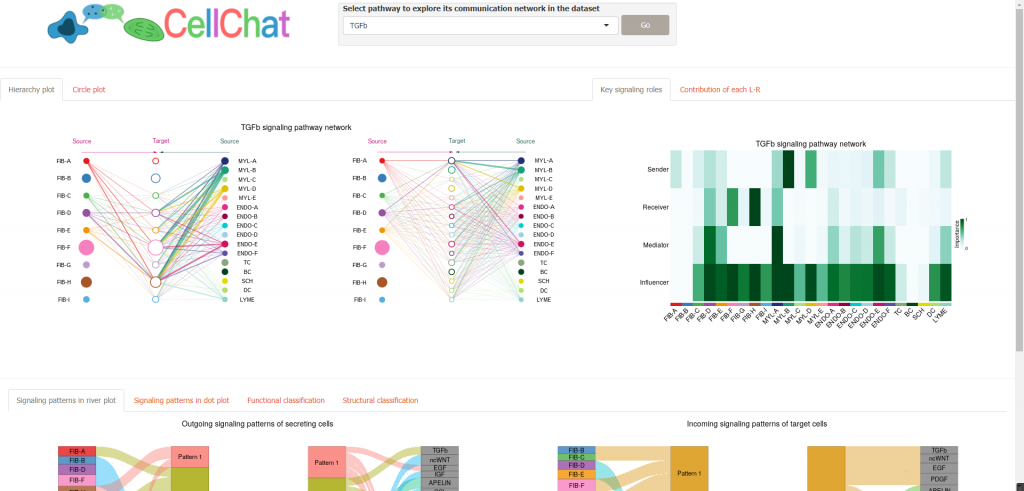
注意:可以用saveRDS(cellchat, file = "cellchat.rds")来保存rds文件。
参考资料:
1.https://github.com/sqjin/CellChat
2.https://github.com/sqjin/CellChatShiny
3. http://www.cellchat.org/
 浙公网安备 33010802011761号
浙公网安备 33010802011761号