Glimma包使用Vega和htmlwidgets框架构建了一个基于web的交互差异分析流程,提供了可以探索多个维度的交互式MDS图、MA图等。Glimma创建的图可以嵌入到R Markdown中,或导出为独立的HTML文档。
安装:
if (!requireNamespace("BiocManager", quietly = TRUE))
install.packages("BiocManager")
# 安装 ,其中RNAseq123中含本次演示数据集
BiocManager::install(c("Glimma", "limma"))下面以Glimma的主要MDS图和MA做一个简单的介绍(此处参考官方文档,也采用RNAseq123的演示数据,本文以Glimma+limma做演示)。
1)MDS图
library(Glimma)
library(limma)
# 建议安装2.0最新版
dge <- readRDS(system.file("RNAseq123/dge.rds", package = "Glimma"))
# 交互式分析
glimmaMDS(dge)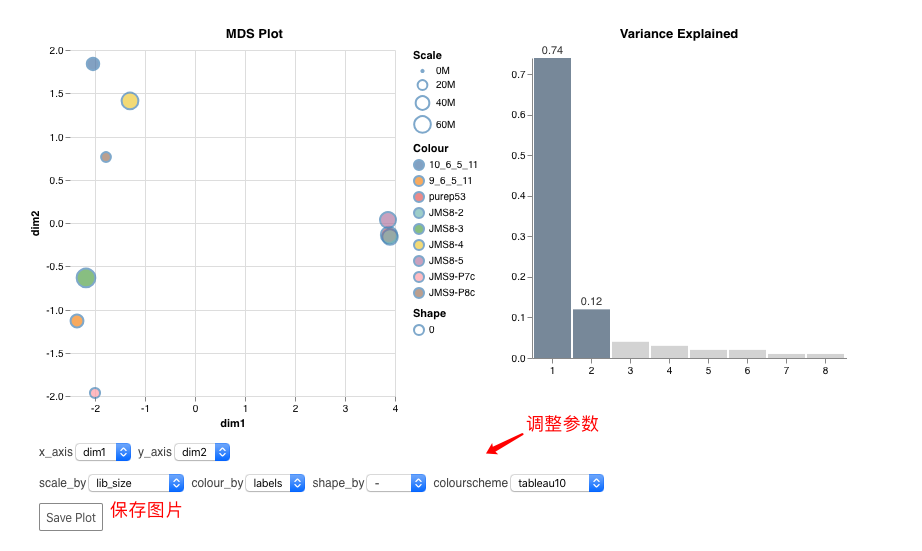
解释:
- 按lib_size或者norm_factors缩放点大小
- 使用colour_by按字段更改点的颜色
- 使用colourscheme更改配色方案
- 使用shape_by更改点的形状
- 将在x轴(x_axis)\(y_axis)更主成分选项
- 使用“Save plot”按钮将图以PNG或SVG格式保存
2)MA图
library(Glimma)
library(limma)
# 演示数据
design <- readRDS(
system.file("RNAseq123/design.rds", package = "Glimma"))
contr.matrix <- readRDS(
system.file("RNAseq123/contr.matrix.rds", package = "Glimma"))
# 用limma做差异分析
v <- voom(dge, design)
vfit <- lmFit(v, design)
vfit <- contrasts.fit(vfit, contrasts = contr.matrix)
efit <- eBayes(vfit)
glimmaMA(efit, dge = dge)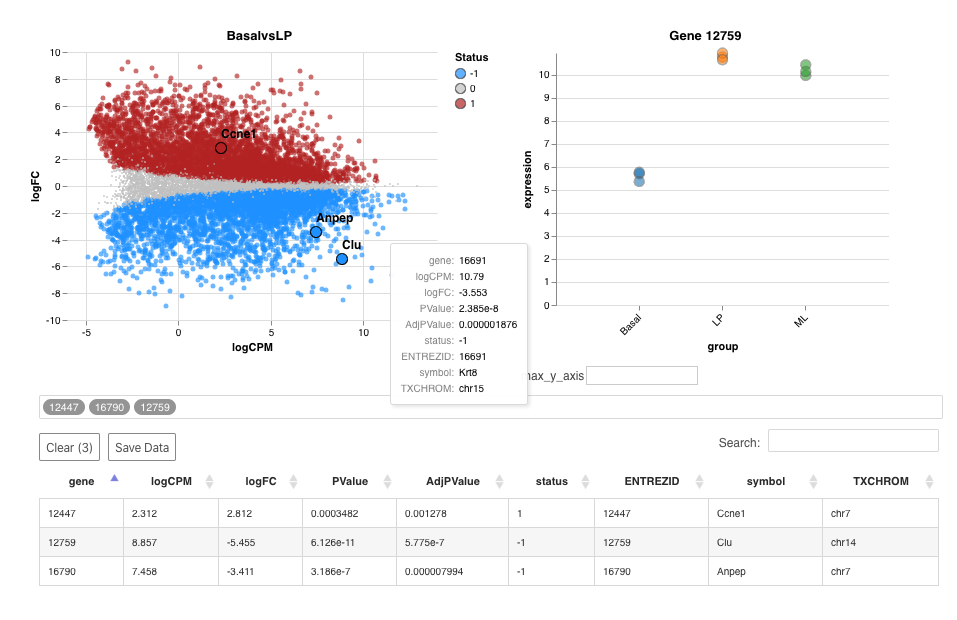
解释:
- 单击摘要图中的点或表中的行以绘制选择的基因表达信息
- 使用搜索框搜索单个基因,搜索结果显示在表中
- 使用max_y_axis字段为表达式图的y轴设置最大值
- 使用“Save Data”下拉列表保存当前选择的基因
- 使用“Save Data”下拉菜单以PNG或SVG格式保存摘要图或基因表达图
Glima对差异分析探索提供了一个便捷的操作界面,总体来说还是非常不错的。
参考资料:
1.http://www.bioconductor.org/packages/release/bioc/vignettes/Glimma/inst/doc/limma_edger.html
2.https://github.com/Shians/Glimma
 浙公网安备 33010802011761号
浙公网安备 33010802011761号