ggmuller由来是作者Rob Noble当时看见 Hermann Joseph Muller 发表了一张关于性别进化优势图(所以作者将这种图形名命为Muller 图)
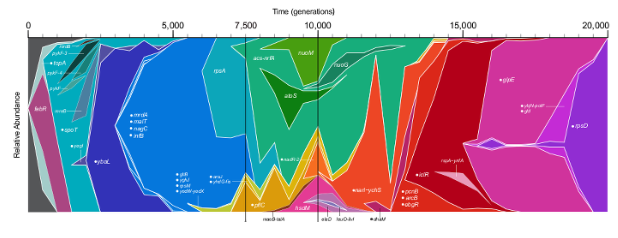
Rob Noble 很感兴趣,于是花了几天时间用ggplot2实现了 Muller 图 的绘制,也就有了我们今天使用的R包。 ggmuller 主要用来绘制种群进化、肿瘤发展等可视化展示。
ggmuller的主要函数为get_Muller_df,需要数据的数据为两个数据框,第一个是文件是包含进化关系的临接矩阵,第二个文件随着时间的变化,对应时间点基因型频率的变化情况, 然后利用函数Muller_plot进行可视化展示。
# 安装正式版
install.packages("ggmuller")
# 安装github开发版
devtools::install_github("robjohnnoble/ggmuller")
# 加载
library(ggmuller)根据作者的文章,整理了一个示例代码,供大家学习:
library(ggmuller)
library(ggplot2)
library(ggthemes)
library(gridExtra)
# 构建进化关系的临接矩阵
edges1 <- data.frame(Parent = 1:3, Identity = 2:4)
# 构建对应时间点基因型频率的变化情况
pop_seq <- function(gens, lambda, start_gen){
c(rep(0, start_gen),
exp(lambda * gens[0:(length(gens) - start_gen)]))
}
# 代数
gens <- 0:150
# 定义基础进化速率
lambda <- 0.1
pop1 <- data.frame(
Generation = rep(gens, 4),
Identity = rep(1:4, each = length(gens)),
Population = c(
1E2 * pop_seq(gens, lambda, 0),
pop_seq(gens, 2 * lambda, 0),
pop_seq(gens, 2 * lambda, 3),
pop_seq(gens, 2 * lambda, 8)
)
)
# 生成Muller图数据框
Muller_df1 <- get_Muller_df(edges1, pop1)
# 自定义颜色
my_palette <- c("white", "skyblue2", "red", "green3")
# 绘图,基于ggpot2,可以在此基础上添加ggplot元素
plot1 <-
Muller_plot(Muller_df1, palette = my_palette) +
theme_few() +
xlim(0, 150) +
scale_x_continuous(expand = c(0, 0)) +
ylim(0, 1) +
scale_y_continuous(expand = c(0, 0))
edges2 <- data.frame(Parent = 1:3,
Identity = 2:4)
pop2 <- data.frame(
Generation = rep(gens, 4),
Identity = rep(1:4, each = length(gens)),
Population = c(
1E2 * pop_seq(gens, lambda, 0),
pop_seq(gens, 2 * lambda, 0),
pop_seq(gens, 4 * lambda, 40),
pop_seq(gens, 8 * lambda, 80)
)
)
Muller_df2 <- get_Muller_df(edges2, pop2)
plot2 <-
Muller_plot(Muller_df2, palette = my_palette) +
theme_few() +
xlim(0, 150) +
scale_x_continuous(expand = c(0, 0))+
ylim(0, 1) +
scale_y_continuous(expand = c(0, 0))
# 组合图片
grid.arrange(plot1, plot2)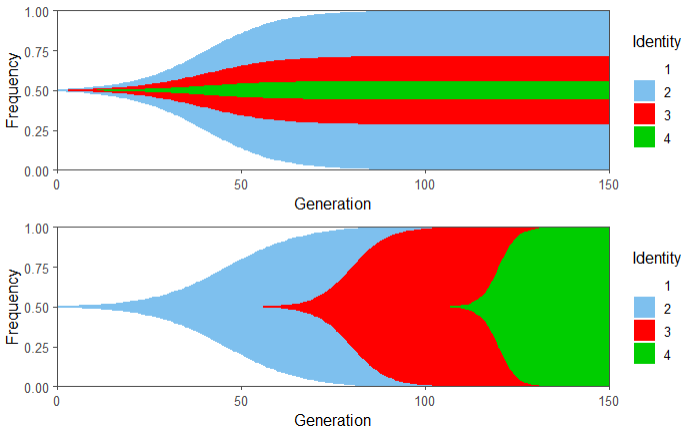
参考资料:
1.https://thesefewlines.wordpress.com/2016/08/20/how-to-ggmuller/
 浙公网安备 33010802011761号
浙公网安备 33010802011761号