在讲到数据分析及其降维,factoextra和FactoMineR是值得推荐的。
这里我们提到了两个R包:i. FactoMineR 包(Sebastien Le, et al., 2008) 用于计算 PCA、 (M)CA、FAMD、MFA 和 HCPC; ii. factoextra 是一个用于多变量数据分析及其可视化的R包。
下面简单介绍factoextra用到的方法:
- 主成分分析(Principal Component Analysis,PCA):用于通过在尽可能的保留重要信息的情况下减少数据的维度来呈现多变量数据中包含的信息(降维)
- 对应分析(Correspondence Analysis,CA):对应分析又称为相应分析,也称R-Q分析,它主要通过分析两个定性变量构成的列联表来揭示变量之间的关系
- 多重对应分析(Multiple Correspondence Analysis,MCA) :指分析多个定性变量构成的列联表来揭示变量之间的关系
- 多因素分析(Multiple Factor Analysis ,MFA ):研究多个因素间的关系及具有这些因素的个体之间的一系列统计分析方法
- 分层多因素分析(Hierarchical Multiple Factor Analysis ,HMFA): 是MFA的扩充,主要针对一些具有层次结构的数据
- 混合数据的因子分析(Factor Analysis of Mixed Data ,FAMD):MFA的一个特例,主要针对一些定性或者定量分析的数据
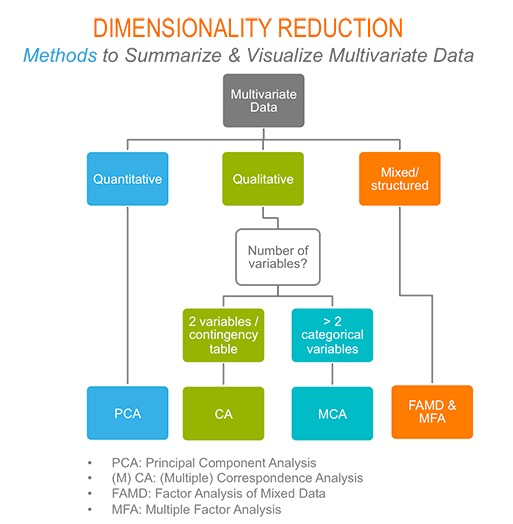
下面是官方的一个方法选择流程,很清晰的给出了各个方法的使用决策:
# 安装R包
install.packages("FactoMineR")
install.packages("factoextra")1)主成分分析
library("factoextra")
library("FactoMineR")
data("decathlon2")
df <- decathlon2[1:23, 1:10]
# 计算主成分
res.pca <- PCA(df, graph = FALSE)
# 特征提取
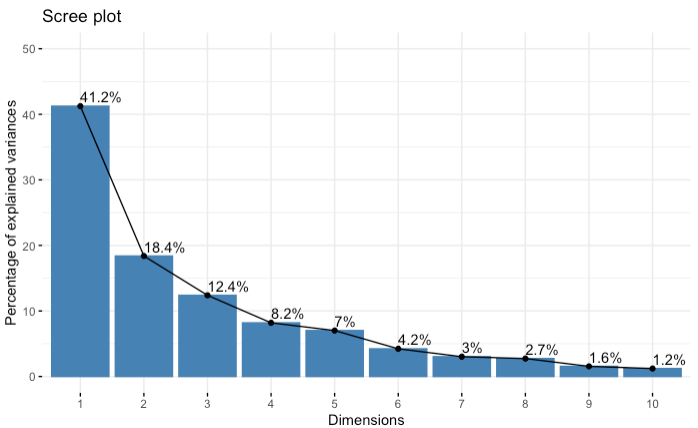
get_eig(res.pca)
# 可视化
fviz_screeplot(res.pca, addlabels = TRUE, ylim = c(0, 50))
# 用颜色变化呈现各个变量的贡献度
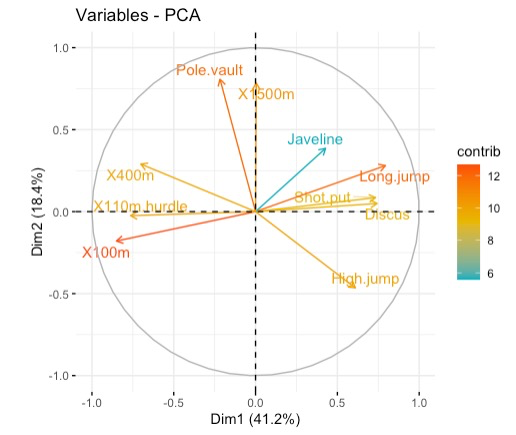
fviz_pca_var(res.pca, col.var="contrib",
gradient.cols = c("#00AFBB", "#E7B800", "#FC4E07"),
repel = TRUE
)
2)对应分析
library("factoextra")
library("FactoMineR")
# 加载数据
data("housetasks")
# 计算 CA
library("FactoMineR")
res.ca <- CA(housetasks, graph = FALSE)
# 可视化
fviz_ca_biplot(res.ca, repel = TRUE)
# 行贡献
fviz_contrib(res.ca, choice ="row", axes = 1)
# 列贡献
fviz_contrib(res.ca, choice ="col", axes = 1)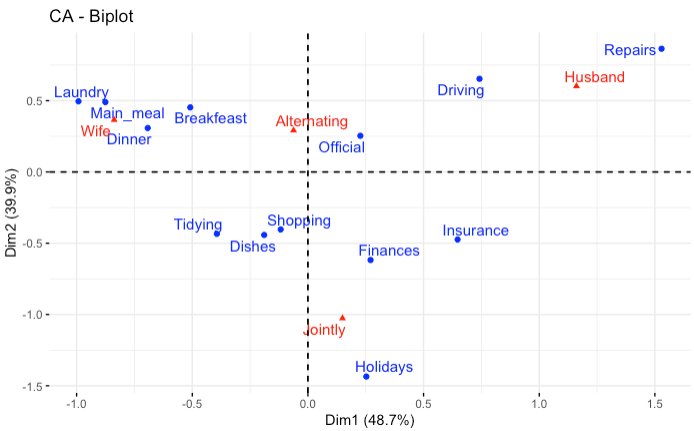
3)多重对应分析
library("factoextra")
library("FactoMineR")
# 加载数据
data(poison)
# 计算MCA
res.mca <- MCA(
poison,
quanti.sup = 1:2,
quali.sup = 3:4,
graph = FALSE
)
grp <- as.factor(poison[, "Vomiting"])
# 绘图
fviz_mca_ind(res.mca,
habillage = grp,
addEllipses = TRUE,
repel = TRUE)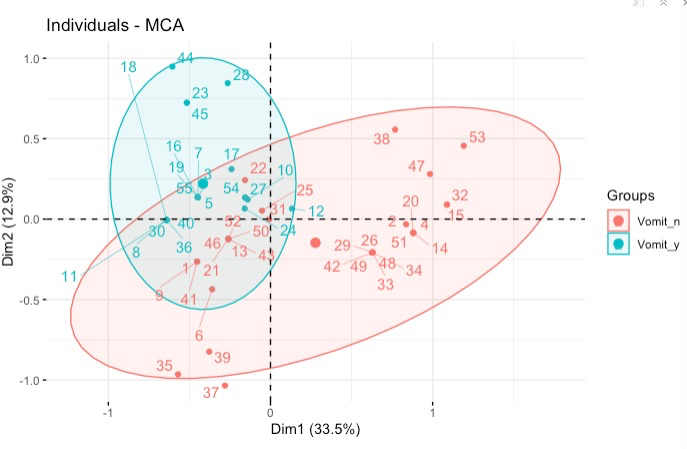
还有FAMD、MFA 和 HCPC等方法可以参考官方文档,基本上如法炮制(如果是拿来主义,原理上可以深度研究FactoMineR包)
factoextra除了上述的分析内容外还包含常见的聚类分析算法:

4)k-means
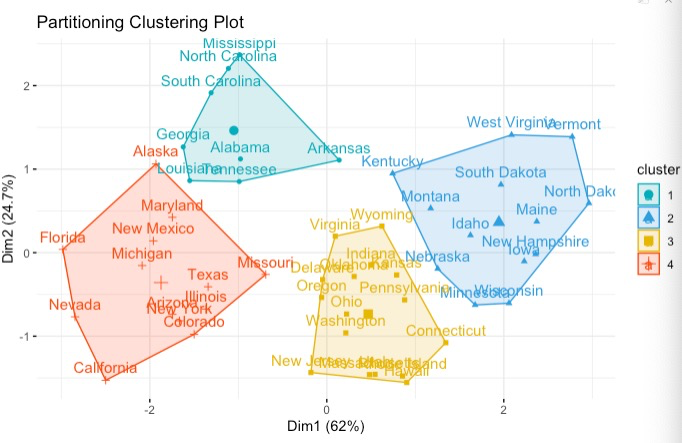
# 演示数据
data("USArrests")
df <- scale(USArrests)
# 计算k-means
set.seed(123)
km.res <- kmeans(scale(USArrests),
4, # 4个中心,简单的理解选择四个初始群
nstart = 25 # 每个中心随机选择多少个数据集
)
# 可视化
fviz_cluster(km.res, data = df,
palette = c("#00AFBB","#2E9FDF", "#E7B800", "#FC4E07"),
ggtheme = theme_minimal(),
main = "Partitioning Clustering Plot"
)
5)层次聚类
# 计算距离
res <- hcut(USArrests, k = 4, stand = TRUE)
# 可视化
fviz_dend(res, rect = TRUE, cex = 0.5,
k_colors = c("#00AFBB","#2E9FDF", "#E7B800", "#FC4E07"))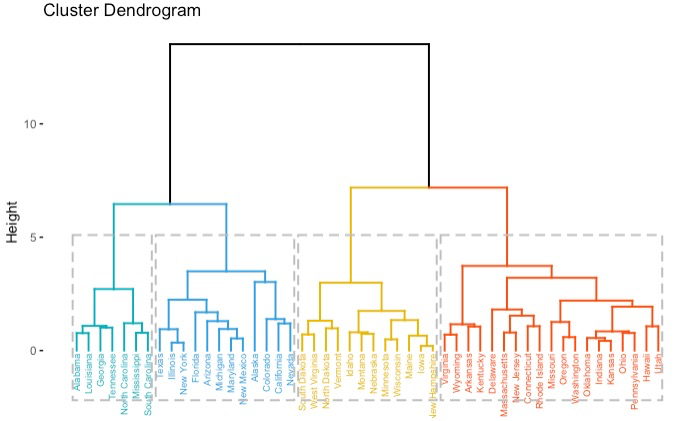
本文主要介绍了其大部分用法(或者称之为应用,原理方面很少提及,主要原因是大部分原理都是一大堆复杂的数学公示,很多生物方向的小伙伴不是感兴趣,而且我写博文的目的主要是给大家提供一个思路,具体的细节,大家可以参考我每篇文章提供的参考资料),如果有堆原理感兴趣的朋友也欢迎留言或者邮件我探讨。
参考资料:
1.https://rpkgs.datanovia.com/factoextra/index.html
 浙公网安备 33010802011761号
浙公网安备 33010802011761号